生命理工学系 News
オーファン酵素遺伝子を探索するための深層学習ベースの 計算手法「DeepES」を開発
オーファン酵素遺伝子の特定による代謝理解と酵素応用に期待
ポイント
- 細菌ゲノムからオーファン酵素遺伝子を探索する深層学習ベースの計算手法「DeepES」を開発した。
- 生合成遺伝子クラスター情報を考慮して酵素機能予測モデルを組み合わせることで、オーファン酵素遺伝子の探索を実現した。
- 酵素遺伝子の探索により代謝の理解や酵素の応用を促進することが期待される。
概要
東京科学大学(Science Tokyo)生命理工学院 生命理工学系の山田拓司准教授(生命理工学コース 主担当)と廣田佳亮大学院生とSalim Felix(サリム・フェリックス)博士らの研究チームは、細菌ゲノムからオーファン酵素[用語1]を効率的に探索するための深層学習ベースの計算手法として「DeepES」を開発しました。
オーファン酵素とは酵素活性は確認されているものの、アミノ酸配列情報が知られていない酵素のことで、酵素データベース上に多数存在しています。オーファン酵素は、アミノ酸配列情報がないためゲノム解析で扱うことが難しく、遺伝子配列と酵素機能の関連の理解を妨げる要因になっています。
そこで本研究では、生合成遺伝子クラスター[用語2]に注目してオーファン酵素遺伝子を探索するための深層学習ベースの計算手法であるDeepES※を開発しました。
そして、DeepESを実際に腸内細菌の4,744種のゲノムに適用することで、236個のオーファン酵素について候補遺伝子を得ることに成功しました。
本研究で開発したDeepESはオーファン酵素遺伝子の探索を目的に設計された初の手法であり、オーファン酵素の候補遺伝子を探索することで代謝の理解や酵素の応用の促進に貢献するものと期待されます。
本成果は、3月5日付の「Bioinformatics」誌に掲載されました。
背景
生物の代謝に必要な生体触媒である酵素については、これまでの研究から明らかになった数多くのアミノ酸配列と機能がデータベースに収録されています。一方で、酵素データベース上には、オーファン酵素[用語1]と呼ばれる酵素活性が確認されているものの、アミノ酸配列情報が未知である酵素が多数存在しています。オーファン酵素は、配列情報がないためゲノム解析で扱うことが難しく、遺伝子配列と酵素機能の関連の理解を妨げる要因になっています。その一方で、オーファン酵素を解明することで、SDGsの実現につながる有用酵素を発見できるのではないかとさまざまな分野で注目が集まっています。
研究成果
本研究では、生合成遺伝子クラスター[用語2]に注目してオーファン酵素遺伝子を探索するための深層学習ベースの計算手法であるDeepESを開発しました。代謝経路 は、さまざまな酵素によって起こる連続反応を記述したものです。そして、これらの酵素をコードする遺伝子はしばしばゲノム上で「生合成クラスター」という形でまとまって存在することが知られています。このような先行研究によって蓄積されてきた知見に基づいて、DeepESは設計されています。
DeepESは、タンパク質言語モデル[用語3]に基づく酵素機能予測モデルを遺伝子配列群に適用し、 それらの予測結果をゲノム上の遺伝子の位置情報と代謝経路上での酵素反応の隣接情報を考慮して評価することで潜在的な生合成遺伝子クラスターを検出します(図)。すなわち、まず入力の遺伝子が目的の酵素機能のいずれかを有しているか予測し、次に目的酵素と予測された遺伝子群がゲノム上で近い位置にコードされているか評価することで、目的の代謝経路に対応する生合成遺伝子クラスターを探索します。
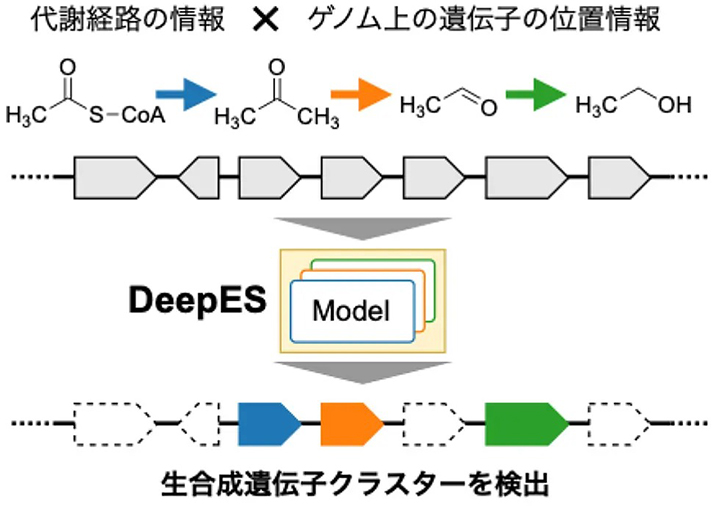
DeepESの概要。DeepESは、代謝経路上で連続して起こる酵素反応の機能の情報とゲノム上で連続して並ぶ遺伝子群の配列の情報を入力して、入力した連続反応に対応する生合成遺伝子クラスターを検出する。
検証の結果から、DeepESの生合成遺伝子クラスターに注目したアプローチの有効性と、オーファン酵素を既存の手法よりも高精度かつ網羅的に検出できる可能性が示唆されました。
そして、DeepESを実際に腸内細菌の4,744種のゲノムに適用した結果、236個のオーファン酵素について候補遺伝子を得ることに成功しました。候補遺伝子が得られたオーファン酵素には、大腸がんなどさまざまな疾患との関連、異物の侵入を防ぐ腸管バリア機能を高めることが知られている短鎖脂肪酸[用語4]の代謝に関わるものも含まれていました。また、最も予測スコアの高い遺伝子をもつ腸内細菌は、先行研究からも類似の代謝を行うことが報告されており、DeepESの信頼性を裏付ける結果が得られました。
社会的インパクト
本手法の開発によってオーファン酵素の候補遺伝子を大規模に探索し、代謝の理解を深められるようになりました。これにより、疾患メカニズムの解明や新規有用分子の発見など、生命科学や医療技術の発展につながることが期待されます。
今後の展開
今後は、DeepESによって予測されたオーファン酵素の候補遺伝子について実験的検証を実施することで、酵素遺伝子を特定すると同時にDeepESの有効性を裏付けられると期待されます。また、特に本研究で扱ったような腸内細菌の重要な酵素の遺伝子を特定することで、腸内細菌研究のさらなる発展および予防医学や治療法の開発に貢献できると考えられます。
- 付記
本研究は、日本学術振興会科研費(16H06279「先進ゲノム解析研究推進プラットフォーム(PAGS)」)により実施されました。
- 用語説明
[用語1] オーファン酵素:酵素活性が確認されている一方で、アミノ酸配列情報が未知である酵素。
[用語2] 生合成遺伝子クラスター:細菌、真菌、植物のゲノムで見られる、代謝プロセスに関連する遺伝子群が集合してコードされている領域。
[用語3] タンパク質言語モデル:アミノ酸を単語、アミノ酸配列を文章とみなし、自然言語処理の技術を応用することで構築された言語モデル。特に近年では、深層学習を用いた大規模モデルが主流である。
[用語4] 短鎖脂肪酸:腸内細菌によって腸管内で産生される主要な最終代謝物質で、数々の疾患との関連のほか腸管バリア機能を高めることが報告されている。
- 論文情報
| 掲載誌: | Bioinformatics |
|---|---|
| タイトル: | DeepES: deep learning-based enzyme screening to identify orphan enzyme genes |
| 著者: | Keisuke Hirota, Felix Salim, Takuji Yamada |
| DOI: | 10.1093/bioinformatics/btaf053 |
研究者プロフィール
山田 拓司 Takuji YAMADA
東京科学大学 生命理工学院 生命理工系 准教授
研究分野:生命情報学、ゲノム・メタゲノム科学関連リンク