生命理工学系 News
【研究室紹介】松浦研究室

主担当 地球生命コース
副担当 生命理工学コース
(地球生命研究所)
教授 松浦友亮![]()
| キーワード | 人工細胞、合成生物学、バイオテクノロジー、膜タンパク質、進化工学 |
|---|---|
| Webサイト | 松浦友亮研究室 |
研究紹介
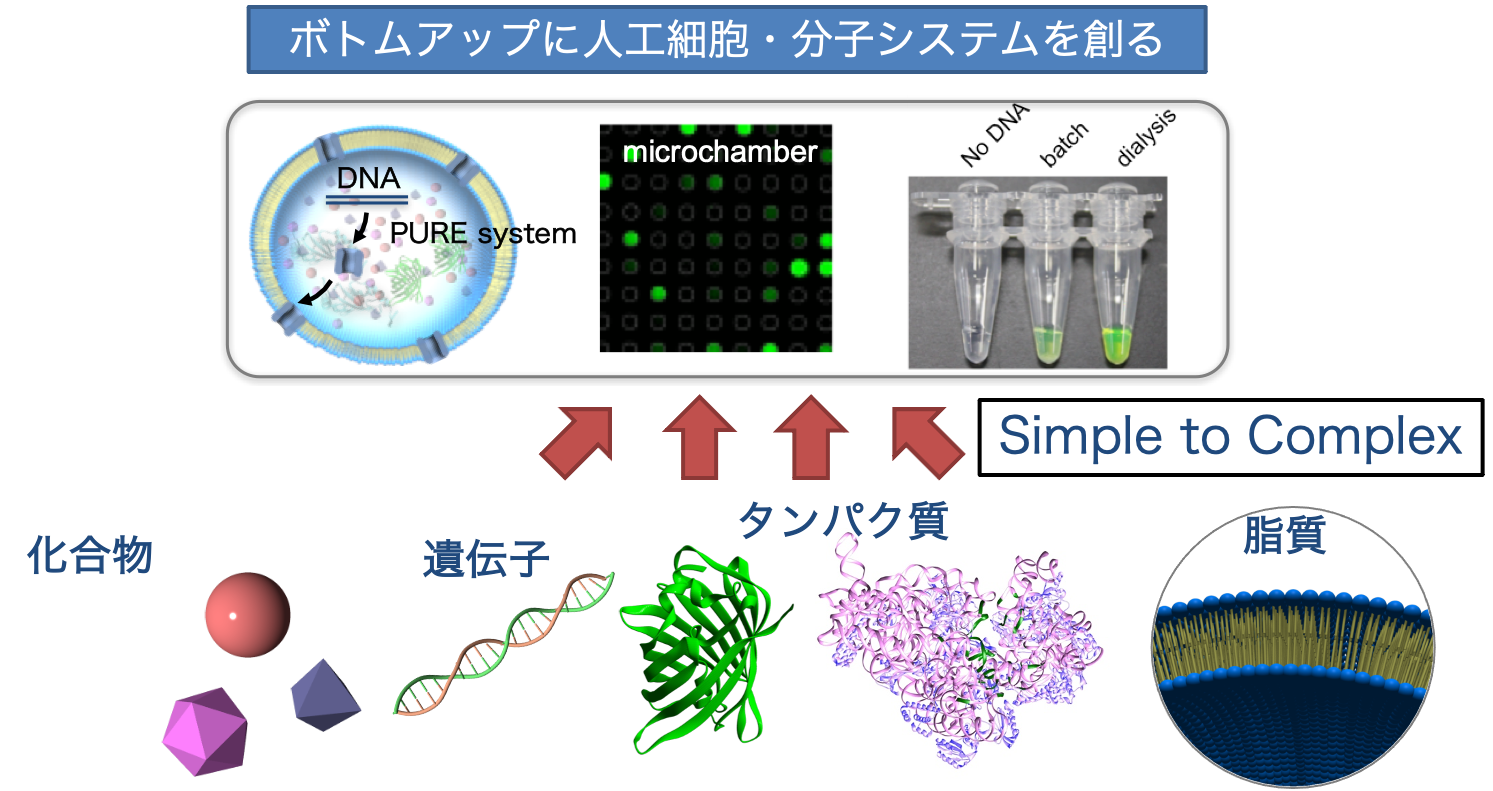
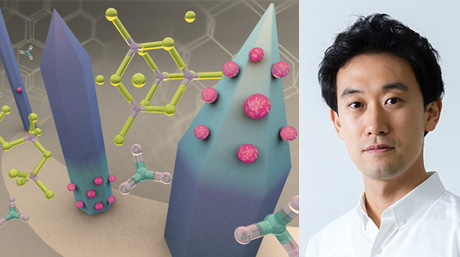
概要:我々のグループでは、生物を構成する部品を組み合わせ、特定の機能に特化した分子システムを作っています。これにより、生命誕生の初期に存在したかもしれない原始細胞の性質を明らかにするとともに、実用化に資する分子や分子システムを創っています【図1,2】。
図1:ボトムアップに人工細胞を創る
図2:動く人工細胞
意義:近年、主として細胞を構成する生体分子を用い、細胞の機能・性質の一部を持つ分子システム(人工細胞など)を構築する研究が盛んに行われています。本研究領域は、2つに大別することができます。
(A)生命の起源及び生命システムの動作原理を理解することを目指す理学的な研究
単純なモデル生物である大腸菌でさえ、数千万以上の分子から構成されてにもかかわらず僅か20分で自らのコピーを正確に作り出すことができます。また、内部状態を柔軟に変化させることで様々な環境条件に適応し生育します。このような驚異的な自己複製能力、環境適応能力がどのようなメカニズムから構成されているのか、また、どのように誕生したのかは、未だ不明な点が多く残されています。自己複製、環境適応能力をもつ人工細胞をボトムアップに作ることができれば、これらの性質を持つ分子システムを構築する十分条件が明らかになります。加えて、非生物から生物を構築するため、非生物と生物の境界が明らかにできることが期待されます。
(B)人工細胞を用いた医療応用、物質生産、化合物の好感度検出などを目指した工学的な研究
人工細胞は、既知の物質のみから構成されているため、構成成分を自由に調整することができます。この利点を生かせば、原理的には人工細胞に望みの機能を付与することが可能です。同様に不要な性質を持たないようにデザインすることも可能です。これは「生きている」ことを絶対条件とする細胞にはない性質です。従って、医療応用、バイオセンサー、物質生産などに資する人工細胞をデザインできることが期待されます。
研究プロジェクト:
人工細胞の構築とその応用
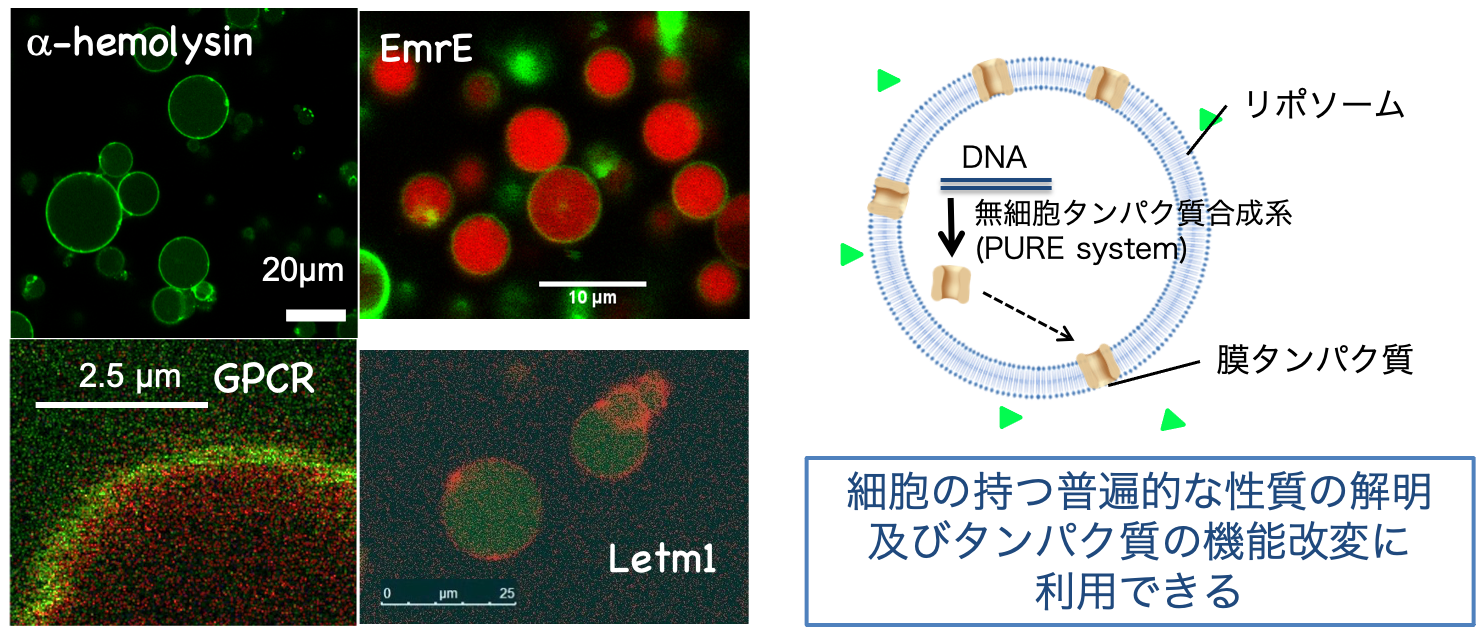
細胞は、その内容物を閉じ込めておくための区画を持っており、その内部で遺伝子からタンパク質を合成します。我々は、この特徴を持った人工細胞を作っています【図3】。この人工細胞は、自分たちで部品から組み立てているので、部品を取り替えたり、追加したりできます。このときに人工細胞の性質を調べることで部品と全体の性質の関係性を明らかにすることができます。また、医療応用、バイオセンサー、物質生産などに資する人工細胞をデザインすることも目指しています。詳しい研究内容は、HPや論文を見てください(ここ!![]() )。
)。
図3:人工細胞の模式図(右)と顕微鏡写真(左)。様々な膜タンパク質を内部で合成した人工細胞を創っている。
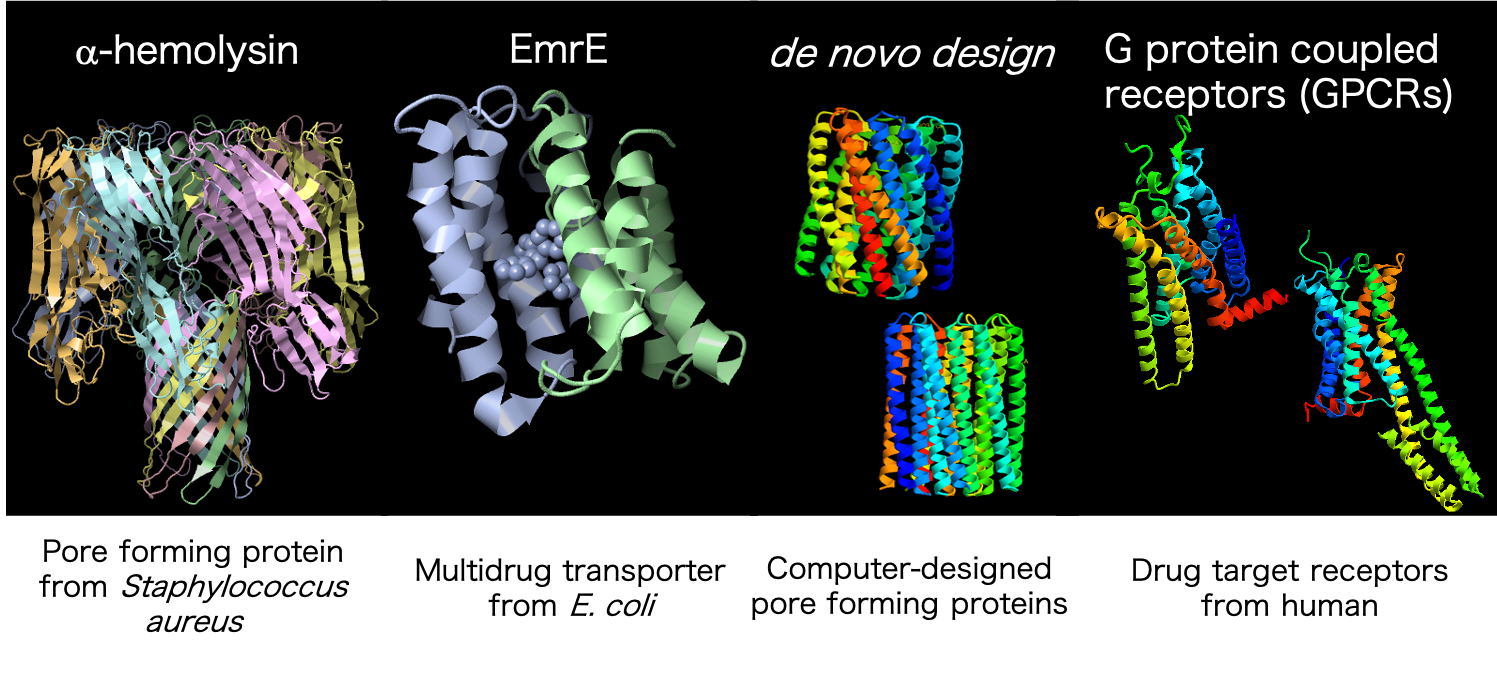
膜タンパク質の進化分子工学
細胞は、その内容物を閉じ込めておくための区画を(細胞膜)持っており、その上に様々な膜タンパク質を持っています【図4】。従って、膜タンパク質は細胞が外部環境を検知するのに最も重要な分子の一つであり、非常に高い感度で細胞外の物質を検出することができます。また、細胞内外の物質を細胞膜を超えて輸送することもできます。このような性質を利用すれば、物質を検出する優れたデバイスができる可能性があります。しかし、天然の膜タンパク質は、人間が使うのに都合が良くできていません。我々は、この膜タンパク質を合成し、その機能を実験室で進化させ、人間が使いやすい分子に作り変える研究を行っています。詳しい研究内容は、HPや論文を見てください(ここ!![]() )。
)。
図4:実験室で進化させている膜タンパク質。バイオセンサーなどに資する分子を創り出すことを目指している。
研究成果
英語論文- 1.Xu, C., Lu, P., Gamal El-Din, T. M., Pei, X. Y., Johnson, M. C., Uyeda, A., Bick, M. J., Xu, Q., Jiang, D., Bai, H., Reggiano, G., Hsia, Y., Brunette, T. J., Dou, J., Ma, D., Lynch, E. M., Boyken, S. E., Huang, P. S., Stewart, L., DiMaio, F., Kollman, J. M., Luisi, B. F., Matsuura, T., Catterall, W. A., and Baker, D. (2020) Computational design of transmembrane pores. Nature 585, 129-134
- 2.Noba, K., Ishikawa, M., Uyeda, A., Watanabe, T., Hohsaka, T., Yoshimoto, S., Matsuura, T., and Hori, K. (2019) Bottom-up Creation of an Artificial Cell Covered with the Adhesive Bacterionanofiber Protein AtaA. J Am Chem Soc 141, 19058-19066
- 3.Dwidar, M., Seike, Y., Kobori, S., Whitaker, C., Matsuura, T., and Yokobayashi, Y. (2019) Programmable Artificial Cells Using Histamine-Responsive Synthetic Riboswitch. J Am Chem Soc 141, 11103-11114
- 4.Matsuura, T., Tanimura, N., Hosoda, K., Yomo, T., and Shimizu, Y. (2017) Reaction dynamics analysis of a reconstituted Escherichia coli protein translation system by computational modeling. Proc Natl Acad Sci U S A 114, E1336-E1344
- 5.Soga, H., Fujii, S., Yomo, T., Kato, Y., Watanabe, H., and Matsuura, T. (2014) In vitro membrane protein synthesis inside cell-sized vesicles reveals the dependence of membrane protein integration on vesicle volume. ACS Synth Biol 3, 372-379
- 6.Kazuta, Y., Matsuura, T., Ichihashi, N., and Yomo, T. (2014) Synthesis of milligram quantities of proteins using a reconstituted in vitro protein synthesis system. J Biosci Bioeng 118, 554-557
- 7.Fujii, S., Matsuura, T., Sunami, T., Nishikawa, T., Kazuta, Y., and Yomo, T. (2014) Liposome display for in vitro selection and evolution of membrane proteins. Nat Protoc 9, 1578-1591
- 8.Fujii, S., Matsuura, T., Sunami, T., Kazuta, Y., and Yomo, T. (2013) In vitro evolution of alpha-hemolysin using a liposome display. Proc Natl Acad Sci U S A 110, 16796-16801
- 9.Matsuura, T., Kazuta, Y., Aita, T., Adachi, J., and Yomo, T. (2009) Quantifying epistatic interactions among the components constituting the protein translation system. Mol Syst Biol 5, 297
- 10.*Kazuta, Y., *Adachi, J., *Matsuura, T., Ono, N., Mori, H., and Yomo, T. (2008) Comprehensive analysis of the effects of Escherichia coli ORFs on protein translation reaction. Mol Cell Proteomics 7, 1530-1540 (*contributed equally)
- 11.Matsuura, T., Yamaguchi, M., Ko-Mitamura, E. P., Shima, Y., Urabe, I., and Yomo, T. (2002) Importance of compartment formation for a self-encoding system. Proc Natl Acad Sci U S A 99, 7514-7517
- 12.Matsuura, T., Miyai, K., Trakulnaleamsai, S., Yomo, T., Shima, Y., Miki, S., Yamamoto, K., and Urabe, I. (1999) Evolutionary molecular engineering by random elongation mutagenesis. Nat Biotechnol 17, 58-61
日本語原稿
- 1.岡村昂典、松浦友亮 (2020) 「リポソームディスプレイ法を用いた膜タンパク質進化分子工学」,膜タンパク質工学ハンドブック,(株)エヌ・ティー・エス, 529-535
- 2.清水義宏、松浦友亮 (2019) タンパク質生合成と数理モデル, 日本生物工学会誌, 97, 492-495
- 3.松浦友亮 (2017) セルフリータンパク質合成系を用いた進化分子工学技術の開発, 日本生物工学会誌, 95, 121-1264
- 4.松浦友亮 (2017) 「6章 環境バイオテクノロジーと環境保全」、ビジュアルバイオテクノロジー(訳書),化学同人,2017, 123-138
- 5.松浦友亮 (2016) ネオバイオ分子創生を目指した配列空間探索, 日本生物工学会誌, 94, 471-472
- 6.松浦友亮 (2015) ジャイアントリポソームを用いたin vitro進化分子工学, 酵素工学ニュース 74,18-21
教員紹介
- 学歴
| 1994年 | 大阪大学工学部 卒業 |
|---|---|
| 1999年 | 大阪大学大学院工学研究科博士課程 修了 博士(工学)を取得 |
- 職歴
| 1999年-2003年 | チューリッヒ大学 博士研究員 |
|---|---|
| 2003年-2006年 | JST さきがけ 代表研究者(兼任) |
| 2003年-2004年 | 大阪大学大学院情報科学研究科助手 |
| 2004年-2006年 | 大阪大学大学院工学研究科コース助手 |
| 2007年-2010年 | 大阪大学大学院情報科学研究科特任准教授 |
| 2010年-2015年 | JST ERATOプロジェクト グループリーダー |
| 2010年-2020年 | 大阪大学大学院工学研究科准教授 |
| 2020年- | 現職 |
- 受賞
| 2016年 | 第52回 日本生物工学会研究奨励賞(斎藤賞) |
|---|---|
| 2015年 | 第4回 大阪大学総長奨励賞(研究部門) |
| 2014年 | 第3回 大阪大学総長奨励賞(研究部門) |
| 2009年 | 酵素工学研究会奨励賞 |
- 所属学会
日本生物工学会、日本生物物理学会、日本蛋白質科学会、日本化学会(生体機能関連部会)
学生へのメッセージ
- 松浦教授より
2020年9月に地球生命研究所にできた研究室です。レゴブロックのように、生物材料をブロックとし人工細胞や分子システムを組み立てる研究を行っています。私のほうが知識・経験が少しばかり多いですが、思考の柔軟性・行動力はみなさんのほうが圧倒的に高いです。「ものづくり」が好きな学生さん、皆さんの持つ力を存分に発揮できる研究環境を整えています。また、国内外の研究者と多くの共同研究を行っています。研究を介して様々な人達と知り合い自由な学生生活を大いに楽しみましょう!
※この内容は掲載日時点の情報です。最新の研究内容については研究室サイト![]() をご覧ください。
をご覧ください。
※ 2025年5月1日:一部最新の情報に更新しました。