生命理工学系 News
【研究室紹介】野澤研究室
生命理工学系にはライフサイエンスとテクノロジーに関連した様々な研究室があり、基礎科学と工学分野の研究のみならず、医学や薬学、農学等、幅広い分野で最先端の研究が活発に展開されています。 研究室紹介シリーズでは、ひとつの研究室にスポットを当てて研究テーマや研究成果を紹介。今回は、ゲノムの立体構造・機能解析に取り組む野澤研究室です。(野澤研究室は2022年4月に発足しました。)

主担当 生命理工学コース
准教授 野澤 佳世![]()
※2022年9月9日に研究室WEBサイトを公開しました。
| キーワード | X線結晶構造解析、クライオ電子顕微鏡解析、ゲノム構造、遺伝子発現制御 |
|---|---|
| WEBサイト | 野澤研究室 |
研究紹介
真核生物のゲノムDNAは高度に折りたたまれ、タンパク質の設計図であるRNAの合成は、そのダイナミックな構造変化によって制御されています。私たちの研究室では、遺伝子のオン・オフを規定する「DNAループ」を中心に、細胞の運命を決定するDNAの折り畳み構造を「原子の世界を覗く技術」で解明したいと考えています。
ヒトは約2万種類のタンパク質遺伝子から構成されていますが、細胞の分化や発生状態に応じたタンパク質発現をコントロールするために、エンハンサーDNAの数は数十万にものぼります。興味深いことにエンハンサーDNAからのタンパク質遺伝子の活性化シグナルは、数キロから 数十Mbpもの長鎖DNAを経て標的遺伝子のプロモーターDNAへと伝わり、この情報伝達は2つのDNA領域が物理的に近接して「DNAループ」構造を作ることによって行われています (Fig. 1)。転写メディエーターは、真核生物特有の転写コファクターであり、この膨大なエンハンサーDNAとプロモーターDNAを対合させ、転写開始点にRNAポリメラーゼIIをリクルートすることで、ほぼすべてのタンパク質発現をコントロールしています。最近では、ガン原遺伝子の上流で多数のDNAループがクラスターを作り、遺伝子の異常な活性化を招くスーパーエンハンサー構造も注目を集めています。スーパーエンハンサー中では、DNAループ同士がクロストークを行うだけでなく、転写メディエーターやRNAポリメラーゼIIに存在する天然変性領域が会合して液-液相分離が起こり、非膜構造の中に転写反応を促進するためのリアクターが形成されることも知られています。
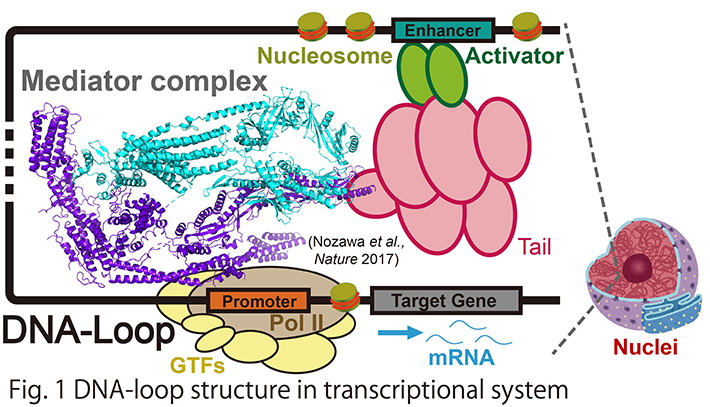
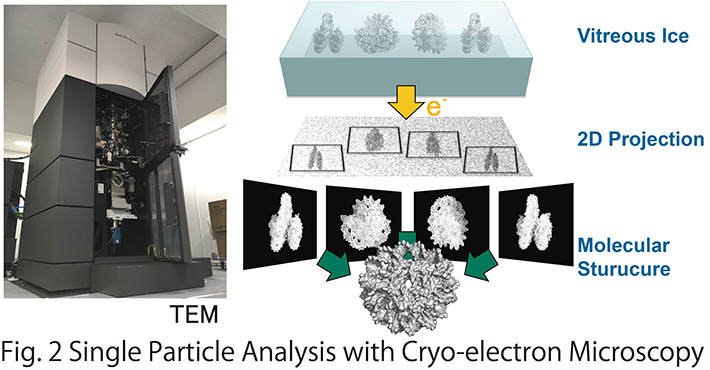
私たちの研究室では、X線結晶構造解析法や2017年にノーベル化学賞を受賞したクライオ電子顕微鏡解析法などの「原子の世界を覗く技術」を用いてゲノム構造とリンクした遺伝子発現制御機構や疾患のメカニズムを解明したいと考えています。特にクライオ電子顕微鏡解析は、氷包埋した分子の複数の溶液構造を一挙に構造解析することも可能であり、得られたスナップショット構造群をつなぎ合わせることで、分子のダイナミクスや酵素反応を一連のムービーのように理解することも可能です (Fig. 2)。最近では、クライオ電子線トモグラフィー法の発展も目覚ましく、凍結細胞のスライスから、原子分解能で構造を決定することも可能になってきました。私たちのグループでは、これまでに酵母の生育に必須の転写メディエーター・15サブユニットのX線結晶構造解析にも成功しています(業績2)。また、クライオ電子顕微鏡解析を通じて新しいクロマチン構造基盤ユニットであるH3-H4オクタソームを独自に発見しました。私たちの研究室では、こうしたゲノムの折り畳み構造を細胞内環境を反映した形で解析するために、電子顕微鏡観察グリット上で、内因性の複合体を精製する技術や核内環境模倣した状態でゲノム構造を観察する技術など、クライオ電子顕微鏡解析の新たな技術プラットフォームの開発にも取り組んでいます。
研究成果
- 原著論文
- 1.M, Nishimura., Y, Arimura., K, Nozawa., H, Kurumizaka.
Linker DNA and histone contributions in nucleosome binding by p53
J Biochem., 168 (6), 669-675 (2020). - 2.K, Nozawa., TR, Schneider., P, Cramer.
Core Mediator structure at 3.4 Å extends model of transcription initiation complex
Nature., 545, 248–251 (2017). - 3.K, Nozawa., R, Ishitani., T, Yoshihisa., M, Sato., F, Arisaka., S, Kanamaru., N, Dohmae., D, Mangroo., B, Senger., HD, Becker., O, Nureki.
Crystal structure of Cex1p reveals the mechanism of tRNA trafficking between nucleus and cytoplasm
Nucl. Acids Res., 41 (6):3901-3914 (2013). - 4.H, Katayama., K, Nozawa., O, Nureki., S, Aimoto., Y, Nakahara., H, Hojo.
Pyrrolysine Analogs as Substrates for Bacterial Pyrrolysyl-tRNA Synthetase in Vitro and in Vivo
Bioscience, Biotechnology, and Biochemistry, 76, 110653-1-4 (2012). - 5.K, Nozawa., P, O'Donoghue., S, Gundllapalli., Y, Araiso., R, Ishitani.,
T, Umehara., D, Söll., O, Nureki.
Pyrrolysyl-tRNA synthetase-tRNA (Pyl) structure reveals the molecular basis of orthogonality
Nature., 457, 1163-1167 (2009). - 6.K, Murakami., M, Stewart., K, Nozawa., K, Tomii., N, Kudou., N, Igarashi., Y, Shirakihara., S, Wakatsuki., T, Yasunaga. & T, Wakabayashi.
Structural basis for tropomyosin overlap in thin (actin) filaments and the generation of a molecular swivel by troponin-T
Proc. Natl. Acad. Sci. U.S.A., 105, 7200-7205 (2008).
- 総説
- 1.野澤 佳世
超分子複合体の立体構造解析を目指したストラタジー~転写メディエーターの X 線結晶構造解析を例として~
蛋白質科学会アーカイブ, 12, e093 (2019) - 2.M, Nishimura., K, Nozawa., H, Kurumizaka.
Crystallographic analysis of the overlapping dinucleosome as a novel chromatin unit
Biophysics and Physicobiology, 12, 251-254 (2018) - 3.K, Nozawa., TR, Schneider., P, Cramer.
Crystal structure of the 15-subunit core Mediator at 3.4 Å extends the structural understanding of transcription
Photon science 2017 (Deutsches Elektronen-Synchrotron Annual reports) http://www.desy.de/about_desy/annual_reports/index_eng.html
- 4.K, Nozawa.
Core Mediator structure extends transcription initiation model
Human Frontier Science Program Awardees' articles 2017
https://www.hfsp.org/hfsp-news-events/core-mediator-structure-extends-transcription-initiation-model
- 5.野澤 佳世
Mediatorの結晶構造から明らかにされた転写開始機構
ライフサイエンス 新着論文レビュー
DOI:10.7875/first.author.2017.046 (2017)
(2017) - 6.C, Plaschka., K, Nozawa., P, Cramer.
Mediator Architecture and RNA Polymerase II Interaction
J Mol Biol., 428 (12), 2569-74 (2016) - 7.K, Nozawa., R, Ishitani., O, Nureki.
Seikagaku., 82, 612-623 (2010)
- 著書
- 1.野澤 佳世、田口 裕之
実験医学別冊「あなたのタンパク質精製大丈夫ですか?」―(2章4節)
羊土社 (2018) - 2.石谷 隆一郎、野澤 佳世、荒磯 裕平
酵素利用技術大系―基礎・解析から改変・高機能化・産業利用まで ―(1章1節) 理工学系出版社(株)エヌ・ティー・エス (2010)
教員紹介
- 学歴
| 2007年3月 | 帝京大学 理工学部卒業 |
|---|---|
| 2009年3月 | 東京工業大学 生命理工学研究科 修士課程修了 |
| 2012年3月 | 東京大学大学院 理学系研究科 博士課程修了 博士(理学)を取得 |
- 職歴
| 2009年-2012年 | 日本学術振興会 特別研究員DC1(東京大学大学院) |
|---|---|
| 2012年-2012年 | 東京大学大学院 博士研究員 |
| 2012年-2014年 | Human Frontier Science Program Long-Term Fellow(ミュンヘン大学) |
| 2014年-2017年 | マックス・プランク生物物理化学研究所 博士研究員 |
| 2018年-2018年 | 早稲田大学 理工学術院 次席研究員 |
| 2018年-2022年 | 東京大学 定量生命科学研究所 助教 |
| 2022年- | 現職 |
- 受賞
| 2021年 | 日本生化学会 JB論文賞受賞(連名受賞) |
|---|---|
| 2018年 | 日本蛋白質科学会 若手奨励賞優秀賞受賞 |
| 2012年 | 東京大学大学院 理学系研究科 研究奨励賞受賞 |
| 2011年 | 栃木県宇都宮市 市民賞受賞 |
| 2010年 | 第5回 ロレアル-ユネスコ女性科学者 日本奨励賞受賞 |
- 所属学会
日本蛋白質科学会
学生へのメッセージ
体を構成する細胞が同一のゲノムDNA配列を持ちながら、発生・分化の過程で異なった組織を形成できるのは、DNA折り畳み構造が細胞の運命を決定しているからです。このゲノム構造の破綻は、がん、生活習慣病、精神疾患など様々な病気を引き起こします。X線結晶構造解析やクライオ電子顕微鏡解析は、1 mmの百万分の一の世界を可視化し、分子のわずかな構造変化が生命現象の引き金になっていることを実感できるエキサイティングな手法であると同時に、分子標的薬を作るための膨大な情報を与えてくれます。構造決定までには試行錯誤が必要ですが、自分の手で世界初の構造を解き明かして、その構造から新しい分子を設計する面白さを一緒に体感してみませんか?
※この内容は掲載日時点の情報です。最新の研究内容については研究室サイト![]() をご覧ください。
をご覧ください。
※ 2025年5月1日:一部最新の情報に更新しました。