生命理工学系 News
エピゲノムの制御を受けた転写の方程式
エピゲノム異常の影響を反応素過程ごとに理解する
概要
理化学研究所(理研)生命機能科学研究センターエピジェネティクス制御研究チームの梅原崇史チームリーダー、若森昌聡技師、東京大学大学院薬学系研究科の岡部弘基助教、東京工業大学情報理工学院の瀧ノ上正浩准教授(生命理工学コース副担当)、千葉大学大学院理学研究院の浦聖恵教授らの共同研究グループは、細胞の機能発揮に関わる「エピゲノム[用語1]」の化学修飾[用語2]が遺伝子の転写[用語3a]に与える影響を、反応の素過程に分けて定量化する方程式を導出しました。
本研究成果は、がん細胞などで見られるエピゲノム異常が転写のどの反応素過程に影響するかの理解につながると期待できます。 エピゲノムは、ゲノムDNA中の遺伝子をRNAに転写する度合いを制御する、ゲノムDNAの可逆的な化学修飾の仕組みです。細胞核に収められたゲノムDNAはクロマチン[用語4]と呼ばれる凝縮した構造をとっており、エピゲノムは転写前にその凝縮を解きほぐすなど、クロマチン構造を変換する複数の反応素過程を制御します。エピゲノムを特徴づけるヒストン[用語5]の化学修飾には複数のパターンがありますが、それらが転写のどの素過程をどの程度制御するのかはよく分かっていませんでした。
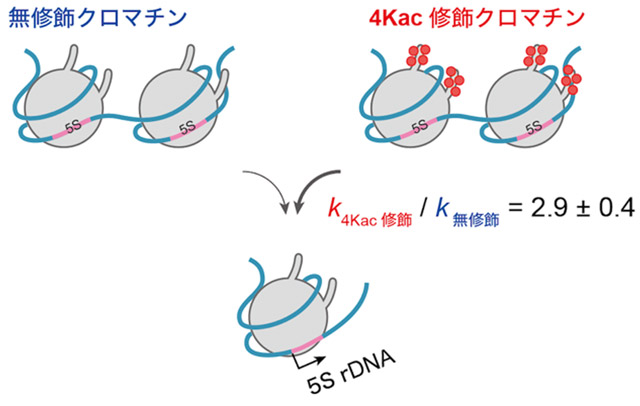
今回、共同研究グループは、5SリボソームDNA(rDNA)[用語6]という短い遺伝子をクロマチンのモデルとして、生化学(作る)・生物物理学(測る)・数理解析(モデル)の異分野融合研究を行い、エピゲノムの制御を受けた転写の素過程ごとの反応速度を求める方程式を導出しました。その結果、今回の実験系では、ヒストンH4のN末端テイルが最も高度にアセチル化された状態[用語7]における転写可能なクロマチンの形成速度は、全くアセチル化されていない状態と比べて2.9倍速いことを見いだしました。
本研究は、科学雑誌『Nucleic Acids Research』のオンライン版(11月26日付:日本時間11月26日)に掲載されました。
エピゲノム修飾が影響する転写反応素過程の理解
背景
「エピゲノム」は、ゲノムDNA中の遺伝子がRNAに転写される度合いを制御する、ゲノムDNAの可逆的な化学修飾の仕組みです。ヒトを含む真核生物の細胞核内では、ゲノムDNAはヒストンタンパク質に巻き付いたヌクレオソーム[用語8]という構造を単位として、たくさんのヌクレオソームがつながった「クロマチン」と呼ばれる凝縮体を作ります。クロマチン中のヒストンとDNAは、ゲノムの場所に応じてさまざまな化学修飾を受け、それらが総体として集積することでエピゲノムが作られます。
エピゲノムを特徴づける代表的な化学修飾は、ヒストンに対するアセチル化です。特に、ヒストンを形成する4種類のタンパク質のうち、ヒストンH4のN末端テイルのリシン残基がアセチル化を受けると、RNA ポリメラーゼ[用語3b]によるクロマチンからの遺伝子の転写が促進されます。この転写の促進は、アセチル化によって転写開始部位付近に位置するヌクレオソームのリモデリングや再配置が促進されることで起こると考えられていますが、そのメカニズムについては不明な点がまだ数多くあります。
細胞内のクロマチンにおいて、ヒストンH4 のアセチル化のパターンには数多くの種類があります。そのうち、N末端から数えて5番目、8番目、12番目、16番目の4カ所のリシン残基(1文字略称はK)が同時にアセチル化された状態(4Kac修飾)は特に重要で、この4Kac修飾は転写が可能なクロマチン領域によく見られます。しかし、クロマチンからの転写は複数の段階的な「反応素過程」で構成されており、その各反応素過程における特定のエピゲノム修飾の貢献度を定量的に分析することは、これまで困難でした。それは、特定の化学修飾を含んだエピゲノムやクロマチンを試験管内で精密に再構成することが難しかったからです。
ヒトゲノムには約2万種類の遺伝子が含まれていますが、これらはRNA ポリメラーゼI、II、IIIのいずれかのRNA合成酵素によって転写されます。遺伝子の中でも、RNA ポリメラーゼIIIによって転写される5SリボソームDNA(5S rDNA)と呼ばれる遺伝子は、転写される塩基配列の長さが約120塩基と短く、遺伝子の本体部分にヌクレオソームを1個だけ含むクロマチンを作るという特徴があります。そのため、5S rDNAは、クロマチンを鋳型とする転写反応を試験管内で分析するモデル実験系として知られています。5S rDNA遺伝子を直列に2つ連結したジヌクレオソームDNAは、アセチル化されたヒストン8量体を用いて再構成した場合、転写可能な状態になります。しかし、試験管内で観察する転写反応は通常、標識されたリボヌクレオチド[用語3c]を用いて転写したRNAを反応後に電気泳動で検出することが多く、その反応の量や進行度を経時的に測定することが困難です。このことも、クロマチンからの転写の素過程におけるエピゲノム修飾の貢献度の定量分析が難しかった理由です。
このような背景に基づき、共同研究グループは、特定のエピゲノム修飾が転写の制御に果たす貢献度を反応素過程ごとに定量的に理解するための方程式を導出することを目指しました。
研究手法と成果
共同研究グループはまず、クロマチンからの転写反応を経時的に測定するための鋳型となるDNAを設計しました。これは、5S rDNA遺伝子に外来遺伝子(c-fos)由来の塩基配列を挿入し、この遺伝子カセットを直列に2つ連結してジヌクレオソームDNAとするものです(図1A)。このジヌクレオソームDNAから転写されたRNAは、蛍光標識した外来遺伝子プローブを用いることで、蛍光相関分光法[用語9]により経時的な検出が可能になります。
次に、4カ所のリシン残基が同時にアセチル化されたヒストンH4(4Kac修飾H4)を得るため、遺伝暗号の拡張技術[注1]を用いて、5番目、8番目、12番目、16番目 のリシン残基をアセチルリシン残基に置き換えた組換えタンパク質を無細胞タンパク質合成技術[用語10]で作製しました。これに、他の3種類のコアヒストン(H2A、H2B、H3)と前述のジヌクレオソームDNAを合わせて、クロマチンを再構成しました(図1C)。さらに、4Kac修飾H4と比較するため、無修飾H4を含むジヌクレオソームも同様に再構成しました(図1B)。
そして、無修飾H4と4Kac修飾H4を含むジヌクレオソームで、DNAとヒストンの巻き付き状態に違いがあるかを調べるため、DNA消化酵素(エンドヌクレアーゼ)を用いた消化アッセイを行いました。この消化酵素は、ヒストンと巻き付いていない裸のDNAを消化しやすいことが知られていますが、消化アッセイではジヌクレオソームの消化パターンに両者の差は見られませんでした(図1D)。このことから、4Kac修飾H4は無修飾H4の状態と比べて、クロマチンを緩める反応素過程を促進しないことが確認できました。
[注1]2015年5月18日 「DNA情報の変換ルールを人為的に改変」|理化学研究所![]()
![図1. クロマチンからの転写に用いる4Kac修飾ジヌクレオソームの再構成 A)ジヌクレオソーム遺伝子カセットX5S197-F2の模式図。5SリボソームDNA(5S rDNA)遺伝子の転写産物を矢印で、その内部制御領域を四角(box A、IE、box C)で、蛍光相関分光測定のための20塩基の外来遺伝子(c-fos)由来のアンチセンスプローブの部分塩基配列を赤で示す。B)無修飾ヒストンH4で再構成した5S rDNA遺伝子ジヌクレオソームの原子間力顕微鏡像。C)4Kac修飾ヒストンH4で再構成した5S rDNA遺伝子ジヌクレオソームの原子間力顕微鏡像。D)エンドヌクレアーゼ(MNase)を用いたジヌクレオソームの消化アッセイ。[無]のレーンは、無修飾H4を含むジヌクレオソームを消化したDNAの泳動、[4]のレーンは、4Kac修飾H4を含むジヌクレオソームを消化したDNAの電気泳動、「MNase」のヒストグラムは、1マイクログラムDNAあたりのMNase使用量を示す。MNaseで消化しないジヌクレオソームDNA(左から1番目と2番目のレーン)は、ヌクレオソーム 2個分のDNAに相当する424塩基対(bp)のバンドが見られる。MNase量の増加に伴って、MNaseで消化されにくいヌクレオソームのDNAを示す145~147bpのバンドが蓄積し、その後減少していくが、その傾向は無修飾H4と4Kac修飾H4でほぼ同様である。これは、ヌクレオソーム構造によるMNase消化の阻害効果が等しく起きていることを示す。なお、本実験に用いたジヌクレオソームDNAはc-fos由来の配列を含まないため、(A)で示した配列よりやや短い。](/bio/news/files/news_201201u_2.jpg)
図1. クロマチンからの転写に用いる4Kac修飾ジヌクレオソームの再構成
- A)ジヌクレオソーム遺伝子カセットX5S197-F2の模式図。5SリボソームDNA(5S rDNA)遺伝子の転写産物を矢印で、その内部制御領域を四角(box A、IE、box C)で、蛍光相関分光測定のための20塩基の外来遺伝子(c-fos)由来のアンチセンスプローブの部分塩基配列を赤で示す。
- B)無修飾ヒストンH4で再構成した5S rDNA遺伝子ジヌクレオソームの原子間力顕微鏡像。
- C)4Kac修飾ヒストンH4で再構成した5S rDNA遺伝子ジヌクレオソームの原子間力顕微鏡像。
- D)エンドヌクレアーゼ(MNase)を用いたジヌクレオソームの消化アッセイ。[無]のレーンは、無修飾H4を含むジヌクレオソームを消化したDNAの泳動、[4]のレーンは、4Kac修飾H4を含むジヌクレオソームを消化したDNAの電気泳動、「MNase」のヒストグラムは、1マイクログラムDNAあたりのMNase使用量を示す。MNaseで消化しないジヌクレオソームDNA(左から1番目と2番目のレーン)は、ヌクレオソーム 2個分のDNAに相当する424塩基対(bp)のバンドが見られる。MNase量の増加に伴って、MNaseで消化されにくいヌクレオソームのDNAを示す145~147bpのバンドが蓄積し、その後減少していくが、その傾向は無修飾H4と4Kac修飾H4でほぼ同様である。これは、ヌクレオソーム構造によるMNase消化の阻害効果が等しく起きていることを示す。なお、本実験に用いたジヌクレオソームDNAはc-fos由来の配列を含まないため、(A)で示した配列よりやや短い。
次に、蛍光相関分光法を用いた経時的な転写反応の検出系を構築しました。試験管内での転写反応には、5S rDNAの転写に必要なRNA ポリメラーゼIIIを含む転写装置が蓄積されているカエル卵母細胞核抽出物を用いました。この抽出液には、内在性の5S rDNAから転写された5s rRNAも多く含まれますが、本実験で用いたジヌクレオソームDNAはカエルゲノムに存在しない外来遺伝子由来の塩基配列を持つため、そのアンチセンスRNA[用語11]を蛍光プローブとして用いることで区別できます。検出の原理は、蛍光標識したアンチセンスRNAプローブがジヌクレオソームDNAから転写されたRNAとハイブリダイゼーション[用語12]すると、蛍光相関分光法によって観察される蛍光標識体の拡散速度が低下することを利用するものです(図2A, B)。検証実験では、c-fos由来塩基配列を含む5S rDNAを転写した場合でのみ、蛍光標識体の拡散速度が低下し(図2C)、さらに転写されたRNAの濃度と拡散速度の相関関係から、RNAの定量が可能であることが確認できました(図2D)。
このように経時的な転写反応の検出系を確立したことで、さまざまな状態のDNAからの転写がどのような速度で進むかを解析することが可能となりました。本実験では、裸のDNA(クロマチン構造をとらないDNA)、無修飾H4で再構成したクロマチン、4Kac修飾H4で再構成したクロマチンの3種類を鋳型として用いて、それぞれの転写データを取得しました(図2E)。
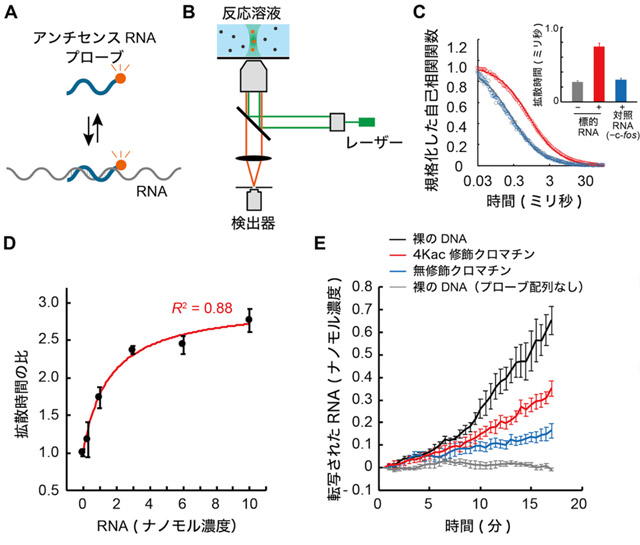
図2. 蛍光相関分光法によるクロマチンからの転写量の経時検出
- A)蛍光標識アンチセンスRNAプローブとRNA転写産物とのハイブリダイゼーションの模式図。
- B)転写されたRNAを検出するための蛍光相関分光法の模式図。転写産物とハイブリダイゼーションしたときのアンチセンスプローブの拡散時間の変化は、1分子レベルでアバランシェフォトダイオードにより検出した。
- C)反応液中の蛍光自己相関関数。5S rDNA遺伝子の2直列コピーからT7 RNAポリメラーゼで末端開始RNAを転写し、鋳型DNAを含まないカエル卵母細胞核抽出液に添加した。丸はデータポイント、実線はフィッティング曲線を示す。挿入図は、アンチセンスプローブの平均拡散時間。赤で示したc-fos由来塩基配列を含む5S rDNAを転写した場合でのみ、蛍光標識体の拡散速度が低下した。
- D)アンチセンスプローブ分子の濃度を関数とする平均拡散時間の検量線。5S rDNA遺伝子の2直列コピーからT7 RNAポリメラーゼで末端開始RNAを転写し、鋳型DNAを含まないカエル卵母細胞核抽出液に添加した。R2は決定係数を示す。これにより、RNAの定量が可能なことが分かった。
- E)5S rRNAの経時検出。使用した鋳型DNAは、灰色:c-fos由来配列を含まない裸の5S rDNA遺伝子、黒色:c-fos由来配列を含む裸の5S rDNA遺伝子、赤色:c-fos由来配列を含む4Kac修飾ジヌクレオソーム5S rDNA遺伝子、青色:c-fos由来配列を含む無修飾ジヌクレオソーム5S rDNA遺伝子を示す。
得られた経時的な転写データをもとに、H4のアセチル化修飾の有無がクロマチンからの転写反応のどの素過程に影響するかを定量的に解析しました。この定量解析を行うため、クロマチンからの転写反応の単純化モデルを構築しました(図3)。このモデルでは、クロマチンからの転写が次の四つの素過程で構成されると仮定しました。
- (I)クロマチンアクセシビリティ
クロマチンへのタンパク質の接近可能性を評価する指標。エンドヌクレアーゼなどの酵素を用いて評価。 - (II)転写可能なクロマチンの形成
RNAポリメラーゼによる転写を可能にするための一連のクロマチン仲介反応をカバーする素過程。 - (III)転写前のプライミング
プロモーター[用語13]上における基本転写因子[用語14]やRNAポリメラーゼによる転写前開始複合体の段階的な分子集合の素過程。 - (IV)RNAポリメラーゼによる転写
RNAポリメラーゼによる転写の素過程。
このモデルでは、裸のDNAからの転写反応は素過程(III)と(IV)で構成され、クロマチンからの転写反応は素過程(I)から(IV)によって段階的に構成されていきます。
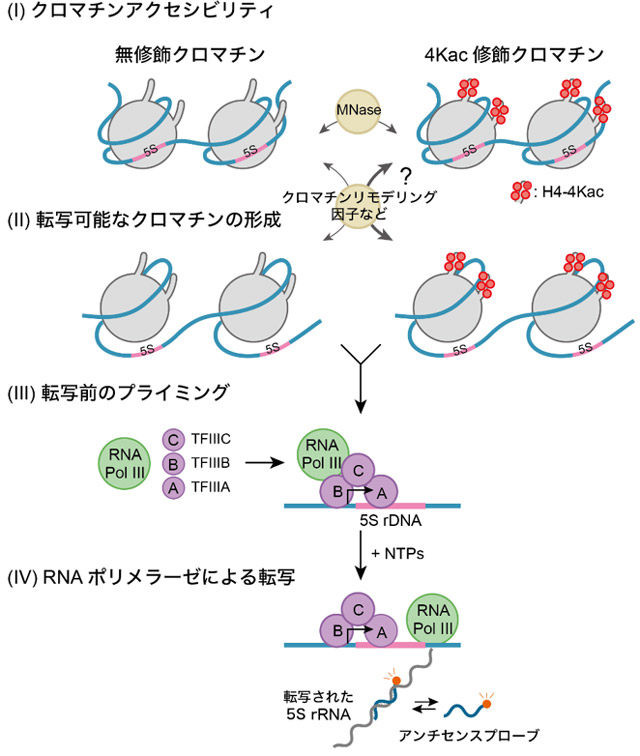
図3. クロマチンからの転写を反応素過程に分けて記述した単純化モデル
(I)から(II)への進行は、生体内ではエピゲノムを認識し、クロマチンを再構成するクロマチンリモデリング因子などが関わる。こうして転写可能になったクロマチンには、RNA ポリメラーゼIII(RNA Pol III)と基本転写因子群(TFIIIA、TFIIIB、TFIIIC)が結合し(III)、RNA ポリメラーゼによる転写が始まる(IV)。
次に、クロマチンからの転写反応の単純化モデルと蛍光相関分光法で得られた転写データに基づいて、転写反応の各素過程の速度論モデルを構築し、転写データを定量化しました。鋳型のDNAや転写産物のRNAの濃度変動を記述する方程式を構築し(図4A)、クロマチンからの転写量Zξと裸のDNAからの転写量Z裸のDNAは、それぞれ図4Bと図4Cの方程式で記述できることを導きました。これらの方程式では、転写反応はミカエリス・メンテン式[用語15]型の酵素反応を仮定し、Z は、反応時刻tにおけるクロマチンまたは裸のDNAからの転写量を示すtの関数となります。
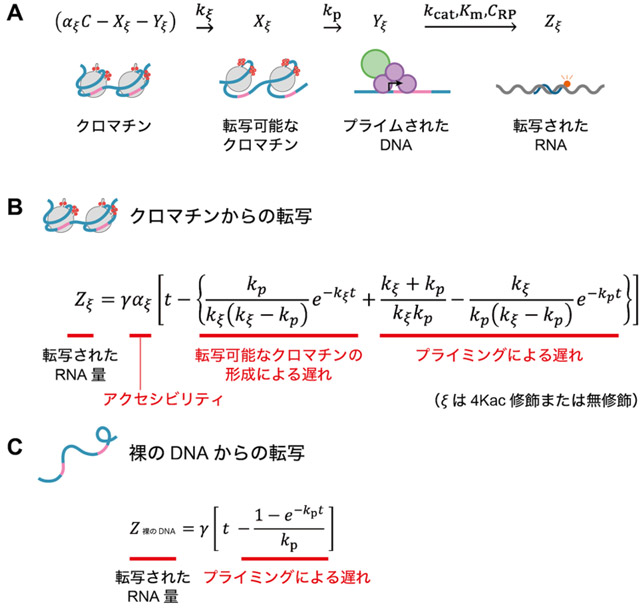
図4. クロマチンからの転写反応の速度論モデル
- A)クロマチンからの転写反応のモデル化の説明。ξは、c-fos由来のアニーリング配列を含むクロマチンテンプレートの種類(4Kac修飾または無修飾のクロマチン)、Cは鋳型の濃度、αξはクロマチン'ξ'のアクセシビリティ、kξはクロマチン'ξ'の転写可能なクロマチンの形成速度、kpはプライミング速度、kcatはRNAポリメラーゼIIIのターンオーバー数、Kmはミカエリス・メンテン定数、CRP はRNAポリメラーゼIIIの濃度を表す。
- B)クロマチンからの転写反応の速度論。γは転写速度、tは反応時刻を表す。
- C)裸のDNAからの転写反応の速度論。
これらの方程式を用いて、素過程(I)から(IV)までのクロマチンからの転写反応の反応速度論をシミュレーションした結果、(II)の転写可能なクロマチンの形成速度kξは、転写量の最終的な増加速度ではなく、転写が始まるまでの時間の遅延に影響することを確認しました。一方、(I)のアクセシビリティαξは、転写が始まるまでの時間の遅延ではなく、転写量の最終的な増加速度に影響することを確認しました。
そこで、今回得られた転写データをこの速度論モデルに当てはめることにより、クロマチンからの転写を制御する速度論パラメータを推定しました。まず、裸のDNAの転写データを速度論の方程式に当てはめ、転写速度γが毎分0.052ナノモル濃度(nM、1 nMは10億分の1モーラー)、およびプライミング速度kpが毎分0.22であることを算出しました(図5A)。
次に、これらの値を用いて、クロマチンからの転写反応の速度論を推定しました。図1Dのエンドヌクレアーゼの消化データに基づき、クロマチンに対するアクセシビリティαξをアセチル化状態によらず1と仮定して、4Kac修飾および無修飾クロマチンの転写データをフィッティングした結果、転写可能なクロマチンの形成速度kξは、4Kac修飾クロマチンの場合は毎分約0.15、無修飾クロマチンの場合は毎分約0.052の値が得られました(図5B)。このフィッティングの全ての解析で、決定係数[用語16]は十分に大きい値(≧0.91)でした。
以上の結果から、本研究で構築した反応速度論モデルがクロマチンからの転写反応を妥当に記述できることが示されました。最終的に、今回の方程式と転写データを用いた速度論分析により、4Kac修飾状態における転写可能なクロマチンの形成速度は、アセチル化が全くない状態と比較して2.9±0.4倍速いことが分かりました(図5C)。
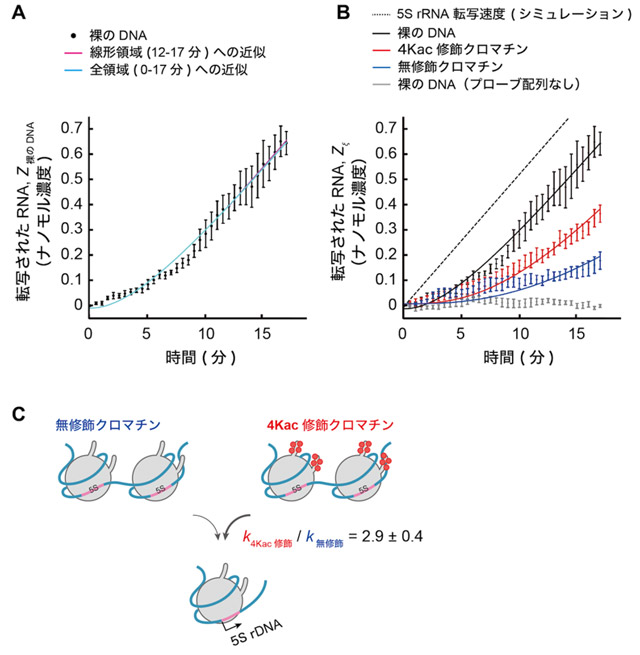
図5. 速度論モデルへのフィッティング解析
- A)転写速度γ値とプライミング速度kp値の決定。γ値は裸のDNAの実験データの線形領域(12~17分)を速度論モデルZ裸のDNA=γt+z1にフィッティングして決定した(z1は切片を示す)。フィット値はγ=0.052±0.003 nM/分、z1=-0.23±0.05 nMであった。得られたγを用いてデータの全領域(0~17分)を速度論モデルZ裸のDNA=γ[t-(1-e-kpt )/kp]+z2でフィットした。z2は初期段階での実験誤差を解消するために導入された切片を示す。フィット値は kp=0.22±0.01/分、z2=-0.010±0.01 nMであった。
- B)転写可能なクロマチンの形成速度kξ値の決定。各DNA状態におけるフィッティング結果。Zξは転写産物の濃度であり、ξは4Kac修飾、無修飾、または裸のDNAを示す。(A)で得られた値をフィッティング解析に用いた。4Kac修飾クロマチンのkξは0.15±0.009/分、無修飾クロマチンのkξは0.052±0.006/分だった。
- C)4Kac修飾によるクロマチンからの転写促進の模式図。Bの結果から、4Kac修飾の状態は、無修飾の状態と比べて転写可能なクロマチンの形成速度kξが2.9±0.4倍速いことが判明した。
今後の期待
本研究で定式化したエピゲノム転写の方程式は、ヒストンのアセチル化修飾の意義の定量的な理解に留まらず、ヒストンやDNAに対するアセチル化以外の化学修飾やその組合せの分析にも広く適用できます。また、細胞の核内においてエピゲノムを鋳型として起こる生体内現象には、転写以外にエピゲノムの複製や修復などがあります。今後、これらの現象においてもそれぞれの反応素過程におけるエピゲノム修飾の貢献度を定量的に分析する研究に応用できると考えられます。
なお、今回再構成した4Kac修飾のクロマチンは、ヒストンH4の5番目と8番目のリシン残基の同時アセチル化修飾を含みます。この同時アセチル化状態は、がん遺伝子の発現を活性化し、アセチル化修飾に結合するタンパク質を阻害する薬剤に対して抵抗性を示すことが分かっています[注2]。がん細胞などで見られるエピゲノムの異常を理解する上でも、エピゲノム修飾がクロマチンからの転写の反応素過程の速度に与える影響を定量化する方程式は重要なツールになると期待できます。
[注2]2018年8月8日「『がんエピゲノム』を検出する新手法」|理化学研究所![]()
共同研究グループ
理化学研究所 生命機能科学研究センター エピジェネティクス制御研究チーム
チームリーダー : 梅原崇史(うめはら たかし)
技師 : 若森昌聡(わかもり まさとし)
東京大学 大学院薬学系研究科
助教 : 岡部弘基(おかべ こうき)
教授 : 船津高志 (ふなつ たかし)
東京工業大学 情報理工学院 情報工学系
准教授 : 瀧ノ上正浩(たきのうえ まさひろ)
千葉大学 大学院理学研究院
教授 : 浦聖恵(うら きよえ)
研究支援
本研究は、科学技術振興機構(JST)戦略的創造研究推進事業さきがけ「細胞機能の構成的な理解と制御(研究総括:上田泰己)」、日本学術振興会(JSPS)科学研究費助成事業基盤研究B「超活性クロマチンの高分解能検出を介した疾患エピゲノム制御機構の理解(研究代表者:梅原崇史)」等による支援を受けて行われました。
- 用語説明
[用語1] エピゲノム : 細胞内の全DNAの塩基配列として記録された遺伝情報の総体を指す「ゲノム」に対し、DNAやヒストンの化学修飾などによって細胞の個性を記憶する情報の総体を「エピゲノム」と呼ぶ。
[用語2] 化学修飾 : 生体分子に限らず、環境中に存在する分子は、環境内の他の分子と反応してさまざまな化学修飾を受けうる。タンパク質では、構成するアミノ酸の側鎖の官能基にアセチル化やメチル化などのさまざまな化学修飾が起こる。アセチル化修飾の場合、リシン残基の側鎖アミンやN末端残基の主鎖アミンにアセチル基(CH3CO−)の結合が起こる。通常、ヒストンの個々のリシン残基の側鎖は、アセチル基転移酵素と脱アセチル化酵素による可逆的な酵素反応を介して、アセチル化修飾があるかないかの2状態のどちらかで存在する。
[用語3a] 転写 : 遺伝子DNAの塩基配列をRNAポリメラーゼが読み取り、2本鎖DNAの片方の塩基配列に対応するRNAを合成する反応を「転写」と呼ぶ。「RNAポリメラーゼ」は、DNAを鋳型としてRNAを構成する基本単位の「リボヌクレオチド」を重合する酵素であり、真核生物は3 種類のRNA ポリメラーゼI、II、IIIを持つ。多くの遺伝子は、その本体部分だけで転写が起こるのではなく、遺伝子の上流または下流に転写反応を質的・量的に変化させるDNA配列を持つ。今回の実験系で用いた5S rDNA遺伝子は、遺伝子の本体部分と重なる位置に内部制御領域と呼ばれる遺伝子発現の制御配列を持ち、RNA ポリメラーゼIIIに認識される。
[用語3b] RNAポリメラーゼ : 転写[用語3a]を参照のこと。
[用語3c] リボヌクレオチド : 転写[用語3a]を参照のこと。
[用語4] クロマチン : ひも状の長鎖からなるゲノムDNAは、部分的にヒストンと複合体を形成して数珠のような繊維状の構造を形成する。この構造をクロマチンと呼ぶ。
[用語5] ヒストン : DNAを巻き付けることで、長大なDNAを核内に納めるタンパク質をヒストンという。代表的なヒストンはH1、H2A、H2B、H3、H4の5種類があり、H2A、H2B、H3、H4の4種類のコアヒストンが2つずつ集まって、ヒストン8量体を形成する。ヒストンH4は、酵母からヒトに至る真核生物のタンパク質で、アミノ酸配列が最も高く保存されている。
[用語6] リボソームDNA(rDNA) : 翻訳装置を構成するリボソームRNA(rRNA)をコードする遺伝子。RNA ポリメラーゼIまたはIIIにより転写され、細胞内のRNAのほとんどを占める。rRNAの種類は沈降定数(S)で命名されており、5S rDNAは5S rRNAをコードする。
[用語7] ヒストンH4のN末端テイルが最も高度にアセチル化された状態 : ヒストンH4のN末端領域は、特定の構造を持たないテイル(尾部)として溶液中で揺らいでいる。ヒストンテイルは、N末端側の1残基目から数えて5番目、8番目、12番目、16番目の4カ所のアミノ酸が塩基性のリシン残基(1文字略称はK)で構成される。この4カ所のリシン残基側鎖のアミノ基が全てアセチル化修飾されたヒストンH4は、細胞内においてN末端テイルが最も高度にアセチル化された状態であり、転写可能なクロマチンによく見られる。
[用語8] ヌクレオソーム : 真核生物の細胞核内において、DNAとヒストン8量体が周期的に巻き付いて形成する複合体をヌクレオソームと呼ぶ。
[用語9] 蛍光相関分光法 : 蛍光分子を含む試料の微小範囲に光を照射して、蛍光強度の揺らぎを介して分子の運動性を1分子レベルで測定する方法。分子の拡散計数やその変化を計測できる。
[用語10] 無細胞タンパク質合成技術 : 大腸菌などの生きた細胞を使用せず、代わりに大腸菌などの各種細胞内に存在する酵素や基質などを利用してタンパク質を合成する方法。転写(DNAからmRNAを合成する段階)と翻訳(mRNAからタンパク質を合成する段階)を同時進行する方法と、翻訳のみを行う方法がある。本研究では、転写と翻訳を同時進行する方法を用いた。
[用語11] アンチセンスRNA : DNAの配列として書かれた遺伝情報はRNAに転写される。タンパク質をコードするメッセンジャーRNA(mRNA)や、tRNA、rRNAのように機能的な配列として転写されたRNAをセンスRNAと呼ぶのに対し、それらの塩基配列に相補的な塩基配列を持つRNAをアンチセンスRNAと呼ぶ。
[用語12] ハイブリダイゼーション : DNAまたはRNAの2本の一本鎖核酸が互いに相補的な塩基配列を持つ場合に、該当配列間の水素結合によって生じる2本鎖の形成をハイブリダイゼーションと呼ぶ。今回の実験系では、蛍光標識したRNA分子プローブが溶液中に存在しており、転写反応によってRNAが生じると、蛍光分子プローブがハイブリダイゼーションして2本鎖のRNA複合体が形成される。この複合体の拡散係数を蛍光相関分光法で計測することにより、転写産物の濃度とその変化を経時的に測定できる。
[用語13] プロモーター : DNA上で遺伝子(RNA)として転写される領域の近くにあり、遺伝子を発現させる機能を持つ塩基配列。5S rDNA遺伝子では、内部制御領域がプロモーターにあたる。
[用語14] 基本転写因子 : 真核生物の転写反応は、酵素活性を持つRNAポリメラーゼのほかに複数のタンパク質がプロモーターに結合する必要がある。このタンパク質を基本転写因子と呼び、RNAポリメラーゼIIIが行う転写の場合はTFIIIA、TFIIIB、TFIIICの3つがある。
[用語15] ミカエリス・メンテン式 : 酵素反応の速度を表す一般的な式。酵素反応の速度 Vの基質依存性は多くの場合、 V= kcat[E0][S]/(Km+[S]) の式で記述でき、この式をミカエリス・メンテン式という。ここで[E0]は全酵素の濃度、[S]は基質の濃度を表す。kcatはターンオーバー数で、単位時間あたりに1個の酵素が行う酵素反応の回数を示す。Kmはミカエリス・メンテン定数で、酵素と基質の結合の強さに対応し、値が小さいほど酵素と基質の結合が強いことを示す。
[用語16] 決定係数 : 実験で得られた数値データの相関関係をある数式で近似したとき、説明変数に対してその数式で予測される目的変数が、実際のデータとどの程度一致しているかを示す指標。0から1の値を取り、1に近いほど高く予測できていることを意味する。
- 論文情報
| 掲載誌 : | Nucleic Acids Research |
|---|---|
| 論文タイトル : | Quantification of the effect of site-specific histone acetylation on chromatin transcription rate |
| 著者 : | Masatoshi Wakamori, Kohki Okabe*, Kiyoe Ura, Takashi Funatsu, Masahiro Takinoue*, and Takashi Umehara* |
| DOI : | 10.1093/nar/gkaa1050 |
- 「DNA液滴」の形成と制御に成功|生命理工学系 News
- 粒子混雑効果による自発的なラチェット輸送に成功|生命理工学系 News
- DNAオリガミによる人工細胞微小カプセルの開発に成功 ―機能をプログラム可能な分子ロボットの開発に期待―|生命理工学系 News
- 骨格で支えられた人工細胞の形成に成功 薬用カプセルや化粧品などの応用に耐える補強が実現|東工大ニュース
- 人工細胞の中でDNAをコンピュータとして使うことに成功 ―生体内で働く分子ロボットの実現に向けて―|東工大ニュース
- 細胞を模倣した微小反応容器のコンピューター制御に成功 ―人工細胞や分子ロボットの開発に期待―|東工大ニュース
- NHK Eテレ「サイエンスZERO」に瀧ノ上正浩准教授と小長谷明彦名誉教授が出演|生命理工学系 News
- 東工大関係者9名が平成29年度科学技術分野の文部科学大臣表彰で「若手科学者賞」を受賞|東工大ニュース
- 平成28年度「東工大の星」支援STAR 採択者決定|東工大ニュース
- 知能システム科学専攻のグループが工学教育賞を受賞 | 東工大ニュース
- 平成25年度「東工大挑戦的研究賞」授賞式挙行 | 東工大ニュース
- 瀧ノ上正浩講師が第2回「明日の象徴」研究者部門を受賞 | 東工大ニュース
- 瀧ノ上研究室
- 研究者詳細情報(STAR Search) - 瀧ノ上正浩 Masahiro Takinoue
- 情報理工学院 情報工学系
- 工学院 システム制御系
- 生命理工学院 生命理工学系
- 理化学研究所 生命機能科学研究センター
- 東京大学大学院 薬学系研究科・薬学部
- 千葉大学 大学院理学研究院・理学部
- 研究成果一覧
お問い合わせ先
東京工業大学 情報理工学院 情報工学系
准教授 瀧ノ上正浩
E-mail : takinoue@c.titech.ac.jp
Tel : 045-924-5680 / Fax : 045-924-5206