生命理工学系 News
【研究室紹介】 小寺研究室(~2018.3)
情報解析技術に基づく大規模オミックスデータ解析
生命理工学系にはライフサイエンスとテクノロジーに関連した様々な研究室があり、基礎科学と工学分野の研究のみならず、医学や薬学、農学等、幅広い分野で最先端の研究が活発に展開されています。
研究室紹介シリーズでは、ひとつの研究室にスポットを当てて研究テーマや研究成果を紹介。今回は、遺伝子系オミックスと化合物系オミックスを結びつける研究を行う、小寺研究室です。
※小寺講師は2018年4月1日、東工大より転出しました。

生命理工学コース
講師 小寺正明
| キーワード | バイオインフォマティクス、ゲノム情報、ケモインフォマティクス、代謝 |
|---|
研究紹介
近年の生命科学研究では、生体のもつあらゆる分子情報を網羅的に計測して分析するオミックス研究が注目されており、この膨大なオミックスデータから生物学的知見を発見するための情報学的手法の開発が進んでいます。しかし、遺伝子系オミックス(ゲノミクス、トランスクリプトミクス、プロテオミクス)と化合物系オミックス(メタボロミクス、グリコミクス)という二種類のオミックスを連携させる研究はまだチャレンジングな課題として残されています。
私達は、ゲノム情報学と化学情報学を融合したケミカルバイオインフォマティクス研究により、以下のテーマを中心に、遺伝子系オミックスと化合物系オミックスを結びつける研究を行っています。
1. 昆虫・植物間軍拡競争の分子機構
植物は昆虫などによる食害を防ぐため、様々な毒(防御物質)を生合成し、昆虫はそれぞれ独自の対抗手段を編み出してきました。私たちは昆虫による対抗手段を担う分子として、シトクロムP450やグリコシルトランスフェラーゼ、ミロシナーゼなど様々な解毒酵素の進化を昆虫の遺伝情報から読み解きます。
2. 遺伝情報と化学情報の統合解析
植物は昆虫による食害から身を守るための毒(防御物質)など様々な物質を合成し、その中には医薬品として用いることのできる有用な物質も含まれます。私たちは、植物の遺伝情報と化学情報の間の関係を解析し統合することで、植物性物質(ファイトケミカル)の合成経路(代謝)を予測可能にし、有用物質生産や機能性農作物栽培などに応用します。
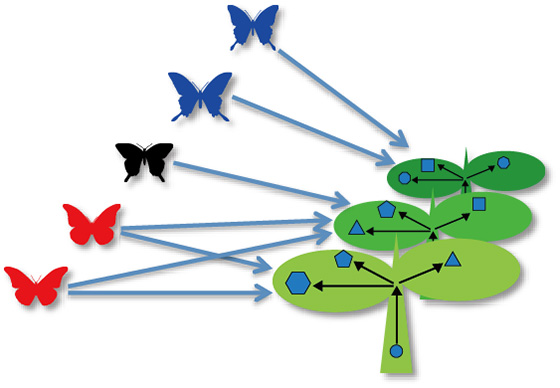
3. 食草選択の進化解析
昆虫・植物間軍拡競争の結果、ある種の草食昆虫は特定の植物種しか食べないという「食草選択」が起こりました。私たちは、日本の全ての蝶とその食草の進化系統関係、およびその遺伝情報と化学情報を統合することで、食草選択の歴史の中で重要な役割を演じたキー・ファクターを発見し、生態系の保全や生物多様性の維持、環境問題解決のヒントを探ります。
研究成果
代表論文
- [1] Yasuo Tabei, Yoshihiro Yamanishi, Masaaki Kotera. Simultaneous prediction of enzyme orthologs from chemical transformation patterns for de novo metabolic pathway reconstruction., Oxford Journals, Vol. 32, No. 12, pp. i278-i287, Jun. 2016.
- [2] Yuki Moriya, Takuji Yamada, Shujiro Okuda, Masaaki Kotera, Zenichi Nakagawa, Toshiaki Tokimatsu, Susumu Goto, Minoru Kanehisa. Identification of Enzyme Genes Using Chemical Structure Alignments of Substrate-Product Pairs, Journal of Chemical Information and Modeling, Vol. 56, No. 3, pp. 510-516, Feb. 2016.
- [3] Iwata Hiroaki, Sawada Ryusuke, Mizutani Sayaka, Kotera Masaaki, Yamanishi Yoshihiro. Large-Scale Prediction of Beneficial Drug Combinations Using Drug Efficacy and Target Profiles, Journal of Chemical Information and Modeling, Vol. 55, No. 12, pp. 2705-2716, Dec. 2015.
- [4] Yoshihiro Yamanishi, Yasuo Tabei, Masaaki Kotera. Metabolome-scale de novo pathway reconstruction using regioisomer-sensitive graph alignments, Bioinformatics, Vol. 31, No. 12, pp. i161-i170, Jun. 2015.
- [5] Masaaki Kotera, Yosuke Nishimura, Zen-ichi Nakagawa, Ai Muto, Yuki Moriya, Shinobu Okamoto, Shuichi Kawashima, Toshiaki Katayama, Toshiaki Tokimatsu, Minoru Kanehisa, Susumu Goto. PIERO ontology for analysis of biochemical transformations: Effective implementation of reaction information in the IUBMB Enzyme List, J Bioinform Comput Biol.12(6):1442001, Dec. 2014.
- [6] Masaaki Kotera, Yasuo Tabei, Yoshihiro Yamanishi, Ai Muto, Yuki Moriya, Toshiaki Tokimatsu, Susumu Goto. Metabolome-scale prediction of intermediate compounds in multistep metabolic pathways with a recursive supervised approach, Bioinformatics, Vol. 30, No. 12, pp. i165-174, Jun. 2014.
- [7] Zhao Jin, Masaaki Kotera, Susumu Goto. Virus proteins similar to human proteins as possible disturbance on human pathways., Syst Synth Biol, Vol. 8, No. 4, pp. 283-295, May. 2014.
国際会議発表(査読有り)
- [1] Masaaki Kotera. Simultaneous prediction of enzyme orthologs from chemical transformation patterns for de novo metabolic pathway reconstruction., The 24th Annual International Conference on Intelligent Systems for Molecular Biology (ISMB 2016), 2016.
- [2] Masaaki Kotera. Metabolome-scale de novo pathway reconstruction using regioisomer-sensitive graph alignments, 23rd Annual International Conference on Intelligent Systems for Molecular Biology, Jun. 2015.
- [3] Masaaki Kotera. PIERO ontology for analysis of biochemical transformations: Effective implementation of reaction information in the IUBMB Enzyme List, GIW / ISCB-Asia 2014, Nov. 2014.
- [4] Masaaki Kotera. Metabolome-scale prediction of intermediate compounds in multistep metabolic pathways with a recursive supervised approach, The 22nd Annual International Conference on Intelligent Systems for Molecular Biology (ISMB 2014), Jun. 2014.
その他の論文・著書など
- [1] Masaaki Kotera, Susumu Goto. Metabolic pathway reconstruction strategies for central metabolism and natural product biosynthesis, Biophysics and Physicobiology, Vol. 13, 195-205, Jul. 2016.
- [2] 小寺正明, 五斗進. 教師付き学習法によるメタボローム規模での多段階代謝経路予測, 生物物理, 日本生物物理学会, Vol. 55, No. 3, pp. 160-163, May. 2015.
- [3] Ryusuke Sawada, Masaaki Kotera, Yoshihiro Yamanishi. Benchmarking a wide range of chemical descriptors for drug-target interaction prediction using a chemogenomic approach., Molecular Informatics, Vol. 33, No. 11-12, pp. 719-731, Nov. 2014.
教員紹介
小寺正明 講師 博士(理学)
| 2005年3月 | 博士号(理学・京都大学)取得 |
|---|---|
| 2005 - 2008年 | ダブリン大学トリニティカレッジ(アイルランド共和国) 博士研究員 |
| 2008 - 2014年 | 京都大学 化学研究所 助教 |
| 2014年2月より | 現職 |
| 2015年 | Oxford Journals-JSBi Prize |
|---|
- 所属学会
- International Society for Computational Biology(ISCB)、日本分子生物学会、日本バイオインフォマティクス学会(JSBi)、日本応用動物昆虫学会
教員からのメッセージ
- 小寺講師より
-
大規模解析技術と情報学的技術の導入で、今後さらに新しい知識を発見できる可能性を秘めています。
決まりきった方法論は、まだありません。
興味のある人はぜひ話を聞きに来て下さい。
※この内容は掲載日時点の情報です。