生命理工学系 News
「並行世界でタイムリープを繰り返す」ことで効率的なシミュレーションを可能にするソフトウエアツールを開発・公開
長時間現象を短時間の計算で容易に観察可能に
要点
- PaCS-MDシミュレーションを容易に実行できるソフトウエアツールPaCS-Toolkitを開発・公開
- Gタンパク質共役型受容体から化合物が解離していく過程のシミュレーションでは1,000億分の1の時間短縮を実現
- PaCS-Toolkitの利用によって、計算による生体分子の働く仕組みといった基礎研究・薬剤の設計や効果の予測などの応用研究を加速することが期待
概要
東京工業大学 生命理工学院 生命理工学系の生澤真司大学院生(研究当時)、堀立樹大学院生、Wijaya Tegar(テガル・ウィジャヤ)大学院生らとTran Phuoc Duy(チャン・フ・ズイ)助教(生命理工学コース 主担当)、北尾彰朗教授(生命理工学コース 主担当)は、多数のMDシミュレーション[用語1]を実行し、上手く行った状態からシミュレーションを再実行するサイクルを繰り返すことで、長時間現象を短時間の計算で観察することができるPaCS-MDシミュレーション[用語2]を容易に実行できるツールキットPaCS-Toolkitを開発し公開した。
PaCS-MDシミュレーションでは、同時並行に実行する複数のシミュレーションで観察された中で、最も上手く行った瞬間を複数選び、それらの瞬間に時間を巻き戻し、少し条件を変えて再度シミュレーションを行う、というサイクルを繰り返す。この操作はSFにおいて「並行世界でタイムリープを繰り返す」ことに例えることができる。PaCS-MDシミュレーションはMSM法[用語3]と組み合わせることで、従来の原子解像度のMDシミュレーションでは観察することができないタンパク質とタンパク質、タンパク質とDNA、タンパク質と薬剤などが結合・解離する過程や生体分子の働きに関わる立体構造変化を、短い計算時間で観察することができるシミュレーション法として注目されている。PaCS-Toolkitはさまざまな計算機へのインストールと効率的なシミュレーションを容易に行えるようにすることで、計算による生体分子の働く仕組みといった基礎研究や、薬剤の設計や効果の予測などの応用研究を加速することが期待できる。PaCS-Toolkitは誰でも利用や改良ができるようにPython3でコード化され、GPLv3ライセンスの下で複数の実行例とともにGitHubから配布されている。
本研究成果は、2024年4月5日(現地時間)に科学誌「The Journal of Physical Chemistry B」の特集号「Recent Advances in Simulation Software and Force Fields」にオンライン公開され、同誌4月18日版に掲載予定である。
背景
MDシミュレーションに代表される分子シミュレーションは、タンパク質・核酸・水・イオンなどからなる分子システムの立体構造やエネルギーが時間に沿って変化していく過程を、原子を最小単位としたモデルを使ってコンピュータ上で観測する計算法で、生体分子が働く仕組みの解明や生体分子に結合して薬として作用する分子の設計などに重要な役割を果たしている。しかし、MDシミュレーションが観察できる時間は高々マイクロ秒からミリ秒程度であり、スーパーコンピュータを使ってもそれより長い時間をかけて起こる現象を観察することはできない。一方、2013年にPaCS-MD法が開発されたことにより、複数のシミュレーションを同時に実行し、その中で予め設定した目標に最も近づいた瞬間を複数探し出してランキングし、ランクが高い瞬間にもどり少し軌道を変えてシミュレーションを再実行するというサイクルを繰り返すことで(図1)、本来はまれにしか起こらない現象の発生確率を飛躍的に向上させることが可能となった。この操作によって、現実には長時間をかけて起こる現象を短いシミュレーション時間の中で観察できる。つまりPaCS-MD法は、通常のMDシミュレーションで観測可能な時間よりもはるかに長い時間で起こる現象を短時間の計算で観察することができる画期的な計算法である。PaCS-MD法で得られた結果をMSM法で解析することで、シミュレーションで得られた大量の断片的な情報を統合して、現象が発生する確率や発生に要する時間などさまざまな量を計算できる。最近の研究から現実には数秒から数時間かかる現象をシミュレーション上の時間では数十ナノ秒程度(ナノ秒は10憶分の1秒)で観測できることが明らかになっている。
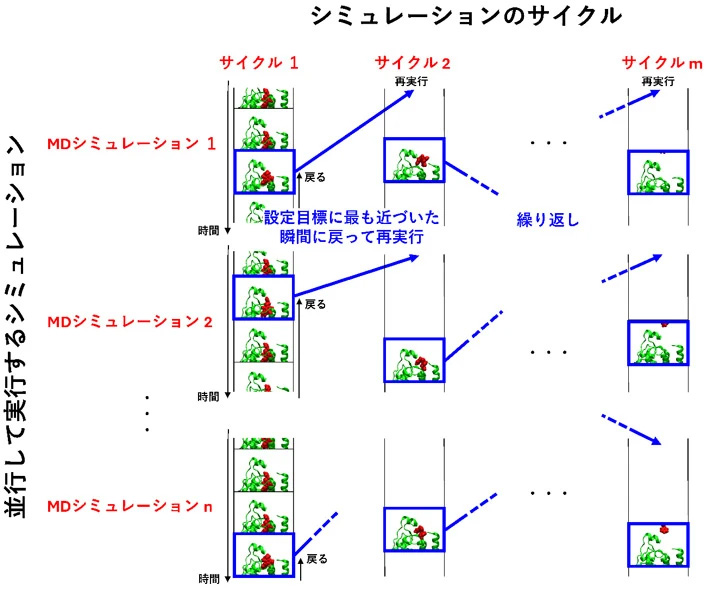
- 図1.PaCS-MDによって観察したい現象が短時間のシミュレーションで観測される様子を表した概念図。並列にn個のMD計算を行うサイクルをm回繰り返す場合を示している。Gタンパク質共役型受容体(緑)から低分子(赤)が解離していく様子を観察した例。2つの分子間の距離がなるべく長くなった瞬間を複数選び、その時間にさかのぼって少し条件を変えてシミュレーションを再実行することで、解離現象が起こる確率を高めている。
研究成果
PaCS-MDシミュレーションを実行するには、異なる条件で複数のMDシミュレーションを実行し、上手く行った状態の情報を抽出するなど、複雑な制御を行うプログラムを作成する必要がある。そこで、本研究においてPaCS-Toolkit の開発・公開を行なった。PaCS-Toolkitはさまざまな種類のPaCS-MDシミュレーションを実行するためのツールが含まれており、TSUBAMEなどのスーパーコンピュータ、グラフィック・プロセッシング・ユニット(GPU)やPCクラスターなどのさまざまな計算機環境で最適化されている。また計算機へのインストールや計算の実行も容易に行えるようにした。
具体的な事例として、多くの医薬品のターゲットとなっているGタンパク質共役型受容体から化合物が解離していく過程を観察した。現実ではこの解離現象に数分を要するが、PaCS-MDではシミュレーション上の時間で3ナノ秒以内に化合物を解離させることができた。これは実際の解離時間の約1,000億分の1にあたる。すなわち解離をMDシミュレーションで観測しようとすると今回の計算の1,000億倍の計算時間が必要であり、不可能である。また結合の強さを示す標準結合自由エネルギーを計算したところ、得られた計算値は実験値とよく一致することも明らかになった。
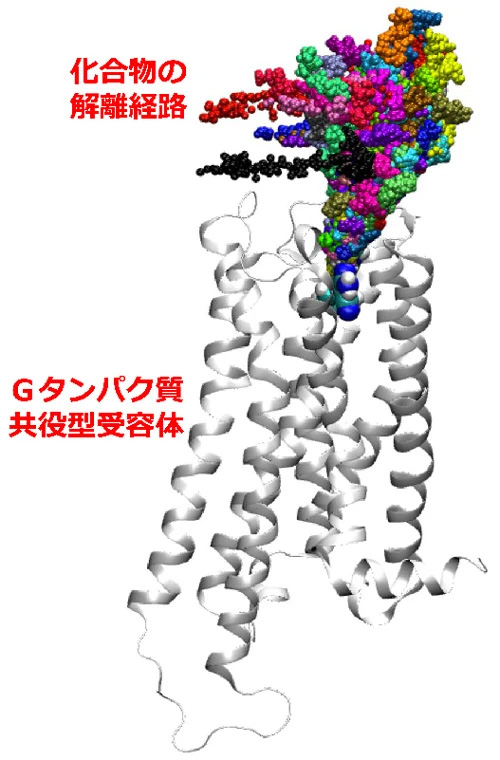
- 図2.Gタンパク質共役型受容体(白)から化合物が離れていく軌跡を、経路ごとに異なる色の球で示したもの。実時間で数分を要する現象を3ナノ秒以下のシミュレーション上の時間で観察することができた。これは解離に要する実時間の1,000憶分の1程度にあたる。
社会的インパクト
PaCS-Toolkitは誰でも利用や改良ができるようにPython3でコード化され、GPLv3ライセンスの下で複数の実行例とともにGitHubから配布されており、同時に提供された入力を使えば、PaCS-MDを簡単に始めることができる。PaCS-Toolkitを使えば、さまざまな計算機へのインストールやPaCS-MDの実行を容易に行うことができるので、計算による生体分子の働く仕組みの解明といった基礎研究や、薬剤の設計や効果の予測などの応用研究を加速することが期待される。
今後の展開
Pythonでプログラミングできるユーザは、PaCS-Toolkitを修正したり、新しいPaCS-MDを実装したりすることもできる。今後はこれまで実装されていなかったPaCS-MDの改良版や、得られた結果を解析するためのツールを追加していく予定である。
- 付記
本研究の計算においては、北尾研究室のPCクラスターに加え、東京工業大学TSUBAME3.0、東京大学物性研究所のスーパーコンピュータ(課題番号:2023-Ca-0019)、自然科学研究機構計算科学研究センターのスーパーコンピュータ(課題番号:23-IMS- C045)、理化学研究所のスーパーコンピュータ「富岳」(課題番号:hp230077, hp230216)の計算資源の提供を受けた。
- 用語説明
[用語1] MD(分子動力学)シミュレーション : 対象とする分子システムを原子の集合体としてコンピュータ上でモデル化し、各原子の運動方程式を数値的に解くことで原子の位置や速度、エネルギーの時間的変化を計算するシミュレーション法。
[用語2] PaCS-MD(並列カスケード選択分子動力学)シミュレーション : 2013年に、原田隆平氏(現在 筑波大学准教授)と北尾が開発したシミュレーション法で、多数の独立なMDシミュレーションを並列に短時間実行し、得られた構造の中から観察したい現象に近い構造を複数選択し、それらを出発構造としてMDシミュレーションを再実行するサイクルを繰り返すことで、現象が起こる実際の時間よりはるかに短いシミュレーション時間で重要な現象を観察できる。
[用語3] MSM(マルコフ状態モデル)法 : 状態間の遷移確率を推定する解析法であり、多数回実行したMDシミュレーションで得られた結果を統合して状態の出現確率、自由エネルギーや現象が起こる時間の計算を可能にし、比較的長時間で起こる現象の性質を推定できる。
- 論文情報
| 掲載誌 : | The Journal of Physical Chemistry B |
|---|---|
| 論文タイトル : | PaCS-Toolkit: Optimized Software Utilities for Parallel Cascade Selection Molecular Dynamics (PaCS-MD) Simulations and Subsequent Analyses |
| 著者 : | Shinji Ikizawa, Tatsuki Hori, Tegar Nurwahyu Wijaya, Hiroshi Kono, Zhen Bai, Tatsuhiro Kimizono, Wenbo Lu, Duy Phuoc Tran, and Akio Kitao |
| DOI : | 10.1021/acs.jpcb.4c01271 |
- がん抑制タンパク質がDNAから解離する過程を可視化|生命理工学系 News
- 新型コロナウイルスの新薬開発につながる候補化合物を発見|生命理工学系 News
- Gタンパク質共役受容体活性化の鍵となる仕組みを解明|生命理工学系 News
- 北尾彰朗 Akio Kitao|研究者検索システム 東京工業大学STARサーチ
- TRAN PHUOC DUY Duy Phuoc Tran|研究者検索システム 東京工業大学STARサーチ
- 北尾研究室
- 【研究室紹介】 北尾研究室|生命理工学系 News
- 生命理工学院 生命理工学系
- 研究成果一覧
お問い合わせ先
東京工業大学 生命理工学院 生命理工学系
教授 北尾彰朗
E-mail : akitao@bio.titech.ac.jp
Tel 03-5734-3373 / Fax 03-5734-3372