生命理工学系 News
ワサビの染色体レベルでのゲノム解読に成功
要点
- 日本を代表する香辛料であるワサビのゲノム配列を染色体レベルにまで繋ぐことに成功し、ハプロタイプレベルで高精度に解読しました
- 本ゲノム情報はワサビの辛味成分の進化機構の解明、栽培化の起源、品種改良など、遺伝資源としての基盤情報の構築に役立つことが期待されます
概要
東京工業大学 生命理工学院 生命理工学系の伊藤武彦教授(生命理工学コース主担当)、田中裕之研究員、堀立樹さん(学士課程4年生、研究当時)、東海国立大学機構 岐阜大学 応用生物科学部の山根京子准教授、山本祥平さん(学部4年生、研究当時)、情報・システム研究機構 国立遺伝学研究所の豊田敦特任教授、東京都立大学 矢野健太郎教授の研究グループは、世界に先駆けてワサビのハプロタイプレベル[用語1]での高精度な全染色体参照ゲノム[用語2]解読に成功しました。
ワサビは日本原産の香辛野菜であり、日本食文化に欠かせない重要な食材です。近年の和食ブームの影響もあり世界的な需要も増加するなかで、抗酸化作用や癌抑制作用など、機能性食品としても注目されています。今回私たちはワサビの全ゲノムの高精度な解読に成功しました。用いた植物材料は、全国わさび品評会において常に上位入賞し、現在最も市場価値が高い品種‘真妻(まづま)’です(図1)。
本研究では、次世代シーケンサー(PacBioおよびIllumina,)とHi-Cとよばれる染色体立体配座捕捉法を用いて染色体スケールのアセンブリを行いました。その結果、ワサビは7本の染色体が4組からなる異質四倍体生物であることを明らかにするとともに、7本x4組=合計28本の染色体から構成される合計1,512.1 Mbの配列データを明らかにしました。どのくらい長い配列をつなげられたかを示す指標であるN50の長さは55.67 Mbでした。さらに、リードマッピングと系統解析により、28本の染色体を2組のサブゲノム、更にはそれぞれを2組のハプロタイプの割り当てに成功しました。3種類の方法(Benchmarking Universal Single-Copy Orthologs、Merqury、Inspector)で評価した結果、得られたゲノム配列は高品質で完全性の高いものであることが示されました。今回明らかとなったゲノム配列は、遺伝や進化などの基礎研究、品種改良など農業分野、さらには在来や野生ワサビの保全のための情報整備など、多くの分野での活用が期待されます。
本研究成果は、日本時間2023年7月11日にNature姉妹誌Scientific Dataのオンライン版で発表されました。

図1. ワサビ品種 ‘真妻’の根茎
背景
ワサビの属するアブラナ科はダイコンやカブ、キャベツやシロガラシなど、経済的にも重要な野菜を数多く含み、すでに90種類のゲノムが解読されています。今回のワサビゲノム解読により、日本固有種ワサビの種分化機構や、辛味成分の生合成にかかわる遺伝的背景の解明も期待されます。
研究成果
アブラナ科植物は過去にゲノム重複を繰り返していることに加え、ワサビは四倍体であり、かつ極めて高いヘテロ性を維持していたこともあり、精度の高いアセンブル[用語3]は非常に困難でした。しかし今回、ロングリードシーケンスやHi-Cなどの最新技術を駆使し、高品質の染色体ゲノム配列の解読に成功しました。
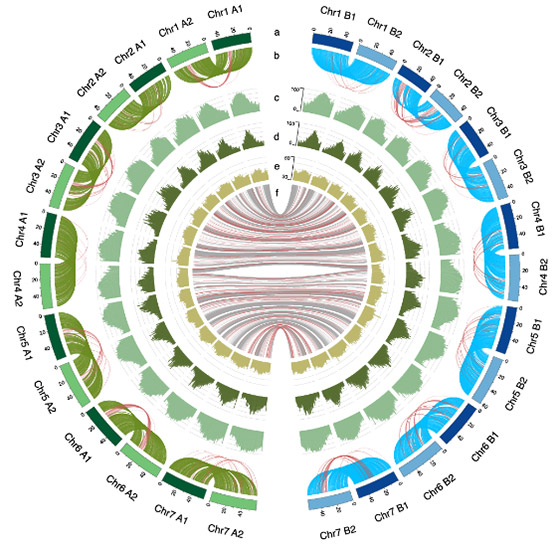
図2. ワサビゲノムの特徴
サブゲノム内染色体および間の遺伝子の並びの保存性
今後の展開
「わさびはなぜ辛い?」などの基本的かつ重要な問いに対し、辛味成分の進化や栽培起源の解明を目指します。また、優良品種の育成に有用なデータを収集し、貴重な品種や在来、野生集団の保全に役立つ情報の整備を行う予定です。
- 付記
本研究は、日本学術振興会科研費(18K05616、16H06279「先進ゲノム解析研究推進プラットフォーム(PAGS)」)により実施されました。
- 用語説明
[用語1] ハプロタイプレベル : (今回用いている意味としては)相同染色体を片親由来ずつに区別して
[用語2] ゲノム : 生物種がもつ遺伝情報の総体
[用語3] アセンブル : 実験で得られた大量のDNA断片配列をつなぎ合わせ、ゲノム配列を復元するコンピュータ処理
- 論文情報
| 掲載誌 : | Scientific Data |
|---|---|
| 論文タイトル : | Haplotype-resolved chromosomal-level assembly of wasabi (Eutrema japonicum ) genome |
| 著者 : | Hiroyuki Tanakaa, Tatsuki Horia, Shohei Yamamotob, Atsushi Toyodac, Kentaro Yanod, Kyoko Yamaneb*, Takehiko Itoha* *責任著者 |
| 研究者所属先 : | a東京工業大学生命理工学院、b岐阜大学応用生物科学部、c情報システム研究機構国立遺伝学研究所、d東京都立大学 |
| DOI : | 10.1038/s41597-023-02356-z |
- 微生物叢中のゲノム配列を長く正確に決定する新手法|生命理工学系 News
- ミドリイガイのゲノム解析からわかった足糸の耐久性の秘密|生命理工学系 News
- 鳥類の性分化に働く遺伝子の共通パターンを発見|生命理工学系 News
- サンゴの天敵・オニヒトデの体表を覆う未知の共在菌をインド・太平洋の広域から発見|生命理工学系 News
- 両親由来のゲノム配列を個別に決定する新手法|生命理工学系 News
- 伊藤武彦 Takehiko Itoh|研究者検索システム 東京工業大学STARサーチ
- 田中裕之 Hiroyuki Tanaka|研究者検索システム 東京工業大学STARサーチ
- 生命理工学院 生命理工学系
- 岐阜大学 応用生物科学部
- 国立遺伝学研究所
- 東京都立大学
- 研究成果一覧
お問い合わせ先
東京工業大学 生命理工学院 生命理工学系
教授 伊藤武彦
E-mail : takehiko@bio.titech.ac.jp
Tel : 03-5734-3430 / Fax : 03-5734-3630