生命理工学系 News
カモノハシとハリモグラの全ゲノム解読に成功!
世界でたった2グループしかいない「卵を産む哺乳類」のゲノムの進化を解明
要点
- 国際共同ゲノムプロジェクトにより「卵を産む哺乳類」のゲノムの特徴を解読。
- 嗅覚とフェロモンに関する遺伝子がカモノハシとハリモグラで大きく異なることを発見。
- 哺乳類がゲノムレベルでどのように進化したかを探る新たな手がかりとなることに期待。
概要
北海道大学大学院地球環境科学研究院の早川卓志助教、東京工業大学生命理工学院生命理工学系の二階堂雅人准教授(生命理工学コース主担当)、株式会社digzymeの鈴木彦有博士、アデレード大学のFrank Grutzner教授、コペンハーゲン大学のYang Zhou研究員、Guojie Zhang教授らの国際共同研究グループは、『単孔類』と呼ばれる「卵を産む哺乳類」であるカモノハシとハリモグラの高精度な全ゲノム塩基配列の決定に成功し、単孔類がどのように進化しているかを明らかにしました。
カモノハシはオーストラリア東部の河川や湖沼に、ハリモグラはオーストラリア全土とパプア島の陸地に生息しています。単孔類はカモノハシとハリモグラの2グループしかおらず、どのように哺乳類が卵を産む爬虫類的な祖先から進化したのかを教えてくれる貴重な存在です。
早川助教ら日本グループは、カモノハシとハリモグラの化学感覚(味覚、嗅覚など)の進化に注目しました。その結果、両種は哺乳類全体でも特別な進化をしており、明確な違いがありました。具体的には、1)ハリモグラは苦味受容体遺伝子がとても少ないこと、2)一方でハリモグラは嗅覚受容体遺伝子を沢山持つこと、3)カモノハシはフェロモン受容体遺伝子を沢山持つことがわかりました。
本研究成果はハリモグラが餌となるアリやシロアリが発する匂いを頼りに餌を探していることや、水中生活者のカモノハシがフェロモンを用いて効率よく仲間とのコミュニケーションや繁殖をしている可能性を示しており、哺乳類のゲノムと生態を結びつける重要な知見です。
なお、本研究成果は、2021年1月6日(水)公開のNature誌にオンライン掲載されました。

池で泳ぐカモノハシ(左)、枯木のシロアリを食べるハリモグラ(右)(写真:早川卓志)
背景
私たちヒトは哺乳類です。多くの哺乳類のお母さんが、お腹の中で胎児を育て、赤ちゃんを産みます。ところが、オーストラリアには、単孔類と呼ばれる「卵を産む哺乳類」が生息しています。この単孔類はカモノハシとハリモグラの2グループしかいません。
カモノハシは水中生活をしており、水鳥のカモのようなクチバシで、水中のエビやザリガニを食べて暮らします。ハリモグラは、地面を掘ったり、アリ塚を壊したりして、土や枯木の中のアリやシロアリを食べて暮らしています。背側に生えた無数の針で身を守っています。カモノハシもハリモグラも、メスは卵を産み、孵化後、母乳で新生児を育てます。
ヒトを含む多くの哺乳類が、なぜ胎生を進化させたのかは、胎生でない生物と比較しなければわかりません。「生きた化石」ともいうべき卵生のカモノハシとハリモグラが生き残っていることは生物学における奇跡です。近年、ゲノム(遺伝子の全て)を解読する技術が急速に進歩し、「哺乳類はどのような祖先で、どう進化したか?」という問いにゲノムの視点で答える機運となりました。そこで、カモノハシとハリモグラの生息国であるオーストラリアと、早川助教ら日本チームを含む計34の国際共同研究機関の研究者による、全ゲノム解読プロジェクトが立ち上がりました。「哺乳類の祖先はどのようなゲノムを持っていて、どう進化したのか」、そして「カモノハシとハリモグラは一体どういう哺乳類で、ゲノムレベルで見るとどう違うのか」という問いに挑戦しました。
研究手法
哺乳類の身体を構成する細胞の1個1個に含まれている『ゲノム』は、20億~30億塩基対のDNA配列からなります。2001年にヒトの全ゲノム配列(ヒトゲノム)が解読されてからは、技術革新に伴い、数多くの哺乳類のゲノムが解読されてきました。カモノハシゲノム自体も、2008年に解読されましたが、当時の技術では決して十分な精度ではありませんでした。
2010年代になると、『次世代シークエンサー』という大量かつスピーディーに塩基配列を決定する装置が普及し、現在までに飛躍的な技術進歩が起こりました。本研究では、カモノハシの全ゲノム配列を最新技術で再決定するとともに、手付かずであったハリモグラの全ゲノム配列も比較相手として新たに決定しました。1分子リアルタイム(SMRT)、10Xリンクドリード、BioNano、Hi-Cといった最新シークエンシング技術を結集して、他に例のない高精度解読に成功しました。
単孔類ゲノムを、ヒトを含む胎生哺乳類のゲノムと比較し、哺乳類がどのように進化したのか、そしてカモノハシとハリモグラがどのように現在まで生き延びて、南半球の生態環境に適応してきたのかを解析しました。
研究成果
全ゲノム配列の決定によって、まず単孔類が胎生哺乳類と進化的にいつ分岐したかを推定できました。哺乳類全体の祖先が、鳥類・爬虫類の祖先と分かれたのは3億50万年前、単孔類と胎生哺乳類の祖先が分かれたのは1億8,760万年前と推定されました。カモノハシとハリモグラが分かれたのは新生代に入ってから(大型恐竜が絶滅してから)で、5,460万年前でした(図1)。
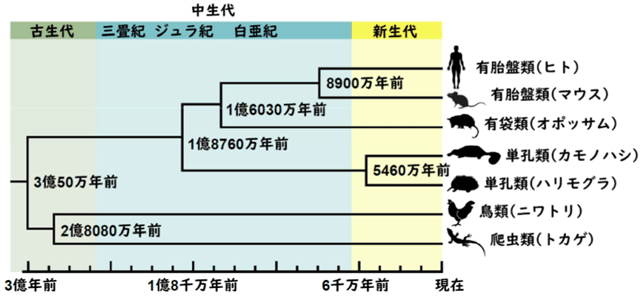
図1. 全ゲノム塩基配列の違いから推定した哺乳類の分岐年代
胎生哺乳類は、性別を決めるX染色体とY染色体を持ちます。しかし、カモノハシは、XとYをそれぞれ5本持ちます。そのため、性染色体の起源は単孔類と胎生哺乳類で異なると考えられています。本研究によって、どのように哺乳類全体の祖先から性染色体が進化したかが明らかになりました。全哺乳類の共通祖先は30対の染色体を持っており、それから156回もの複雑な染色体の再編成を経て、カモノハシが持つ21対の常染色体と5対の性染色体が生じたと推定されました。
独自の生態を進化させたカモノハシとハリモグラの間の違いも明らかにしました。早川助教ら日本チームが注目したのは化学感覚受容体遺伝子です。外界からの化学物質と結合して、「味わい」や「匂い」の電気信号を脳に伝えるタンパク質(化学受容体)を、舌や鼻に作る遺伝子です(図2)。
カモノハシとハリモグラの両種間には予想以上に化学感覚受容体遺伝子の違いがありました。ハリモグラはカモノハシの半分より少ない3個しか苦味受容体遺伝子がありませんでした。一方で、嗅覚受容体遺伝子はカモノハシの倍以上ある693個もありました。これは、ハリモグラが土や枯木の中から、アリやシロアリの発する匂いを手がかりにして探して食べる生態を反映しているものと考えられます。優れた嗅覚を頼りに、一度アリやシロアリのコロニーを見つけてしまえば、あとは長い舌で一網打尽に丸呑みにするだけなので、味覚はほとんど使わないのかもしれません。
他方で、カモノハシはハリモグラがほとんど持っていないフェロモン受容体遺伝子を262個も持っていました。川辺で暮らすカモノハシにとって、効率よく縄張り争いや配偶相手を見つけるために、フェロモンが大きく役立っているのかもしれません。
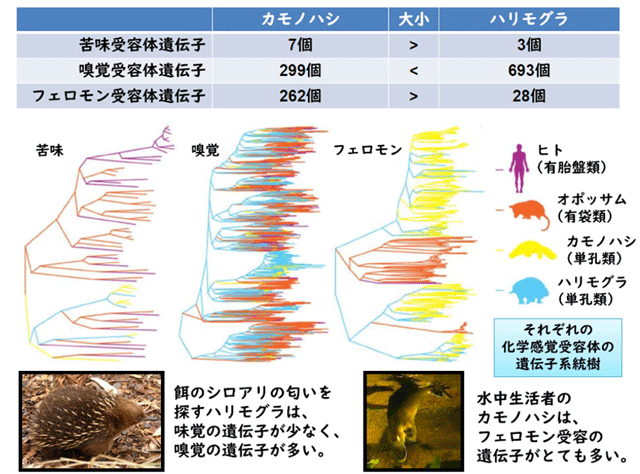
図2. カモノハシとハリモグラの化学感覚受容体遺伝子の個数と進化
本プロジェクトでは、化学感覚以外でも生態とゲノムを繋ぐ多数の発見がされました。例えば単孔類は酸欠に強く、カモノハシの水中生活や、ハリモグラが土を掘ってもぐることを可能にしています。関連して、血中ヘモグロビンに関連する遺伝子の特異的な進化が解析されました。また、カモノハシの大人のオスは、後肢の蹴爪に毒腺を持ち、繁殖期のオス間の闘争に用います。毒のもととなるタンパク質の遺伝子も解析されました。また、卵生や母乳分泌に関する遺伝子も解析され、単孔類と胎生哺乳類がどのように進化したかという、哺乳類進化の根源的な問いに迫る遺伝子の同定が進みました。
今後の展開
哺乳類研究の面白さは、私たちヒト自身も哺乳類であるということです。胎生で、母乳育児をするという、ヒトにとっては当たり前に思う性質は、脊椎動物全体で見ると決して当たり前ではありません。あらゆる生物の全ゲノムを解読する技術がこの数年で急速に進歩し、ヒトから最も遠い、母乳育児はするが卵を産むカモノハシとハリモグラの全ゲノムの解読の成功は、私たちヒトが何者であるかという問いに大きく迫るものです。哺乳類進化のゲノム基盤の解明の研究が躍進するでしょう。
カモノハシもハリモグラも、南半球の一部にしか生息していません。野外での継続的な観察が難しいだけでなく、寿命もカモノハシは約20年、ハリモグラは40年以上と、一生を追うのも容易ではありません。そのため、単孔類の生態は謎が多く残されています。
急速な地球温暖化に伴い、オーストラリアの森林が大規模火災によって減少しており、遺伝的多様性の激減が危惧され始めています。同じ哺乳類の仲間である両種を絶滅の危機から守るためにも、ゲノムレベルでの生態研究は急務であり、本研究成果は重要な土台となることが期待されます。
- 論文情報
| 掲載誌 : | Nature |
|---|---|
| 論文タイトル : | Platypus and echidna genomes reveal mammalian biology and evolution(カモノハシとハリモグラのゲノムが明らかにする哺乳類の生物学的特性と進化) |
| 著者 : | Yang Zhou1、早川卓志2,3、鈴木彦有4、二階堂雅人5、Frank Grutzner6、Guojie Zhang1 (1コペンハーゲン大学生物学専攻、2北海道大学大学院地球環境科学研究院、3公益財団法人日本モンキーセンター、4株式会社digzyme、5東京工業大学生命理工学院、6アデレード大学環境研究所) |
| DOI : | 10.1038/s41586-020-03039-0 |
- オオコウモリ2種の全ゲノム配列を解読|生命理工学系 News
- フェロモン受容機構が退化した哺乳類をゲノム解析で特定|生命理工学系 News
- ほぼ全ての脊椎動物に共通するフェロモン受容体を発見|生命理工学系 News
- アフリカ・シクリッドの多様性は過去のゲノム多型が基盤 ―シクリッド5種の全ゲノム配列を決定して解明―|東工大ニュース
- タンザニア北部にシーラカンスの繁殖集団を発見|東工大ニュース
- 生命理工学の面白さを語る~動画「生命の真理を探り、新たなものを作り出す」~|生命理工学系 News
- 平成27年度「東工大挑戦的研究賞」受賞者決定|東工大ニュース
- 二階堂研究室
- 研究者詳細情報(STAR Search) - 二階堂雅人 Masato Nikaido
- 生命理工学院 生命理工学系
- 北海道大学 大学院 地球環境科学研究院
- 株式会社digzyme
- The University of Adelaide
- University of Copenhagen
- 研究成果一覧
お問い合わせ先
東京工業大学 生命理工学院 生命理工学系
准教授 二階堂雅人
E-mail : mnikaido@bio.titech.ac.jp
北海道大学 大学院地球環境科学研究院
助教 早川卓志
E-mail : hayatak@ees.hokudai.ac.jp
北海道大学 総務企画部 広報課
E-mail : kouhou@jimu.hokudai.ac.jp
Tel : 011-706-2610 / Fax : 011-706-2092