生命理工学系 News
人工進化が生んだ超高親和性RNA–タンパク質ペアの構造基盤解明
RNPツール開発への応用が期待
ポイント
- X線共結晶構造解析により二つの人工RNPの結合様式の違いを原子レベルで解明。
- タンパク質リガンドの結合によって翻訳がONになるセルフリーシステム用リボスイッチを開発。
- γ−ヘモリシンの機能発現を制御する論理回路(AND回路)を構築。
概要
宮崎大学 テニュアトラック推進室 福永圭佑准教授(前東京科学大学特任助教)、九州大学 大学院農学研究院 寺本岳大助教、同角田佳充教授、沖縄科学技術大学院大学(以下OIST) 核酸化学・工学ユニット 横林洋平教授、東京科学大学 地球生命研究所 松浦友亮教授(地球生命コース 主担当)らは、X線結晶構造解析の手法を用いて二つの人工RNA−タンパク質複合体(RNP)の結合様式の違いを解明することに成功しました。さらに、これらRNPを活用した高性能リボスイッチ(遺伝子発現制御用のRNAスイッチ)の開発を行い、無細胞タンパク質合成系(セルフリーシステム)[用語1]においてその性能の高さを実証しました。
CS1−LS4およびCS2−LS12はPD-SELEX法[用語2]を使った実験室での共進化実験により同定された超高親和性・直交性[用語3]のRNA−タンパク質(RBP)ペアです。CS1−LS4、およびCS2−LS12複合体のX線結晶構造解析を行ったところ、これらRNA−RBPペアはユニークな分子認識機構を獲得していたことが明らかとなりました。また、CS1-LS4およびCS2-LS12を分子パーツとして利用し、無細胞タンパク質合成系において翻訳制御が可能なセルフリーリボスイッチを2種類開発しました。このリボスイッチはLS4/LS12が結合することにより翻訳がONになる仕組みで、従来のセルフリーリボスイッチと比較してON/OFF比が大幅に向上していることが特徴です。さらに、リボスイッチを二つ組み合わせることで二成分系の膜タンパク質γヘモリシンの機能発現制御を行うことにも成功しました。この研究成果は、2025年3月23日にOxford University PressのオープンアクセスジャーナルNucleic Acids Researchに掲載されました。
背景
OISTの研究グループはRNA−RBP間の結合様式を改変する手法としてPD-SELEX(Phage Display coupled with Systematic Evolution of Ligands by EXponential enrichment)法を開発していました。合成生物学研究で一般的に分子パーツとして利用されている古細菌由来のL7Aeタンパク質とそれに結合するキンクターンRNAの結合選択性を改変することを目的に進化実験を行い、最終的に直交性を示す二組のRNA−RBPペアCS1−LS4、及びCS2−LS12(解離定数はそれぞれ約7 pM)を取得することに成功しました。一方で、配列相同性の高いこれらRNA−RBPペアがなぜ特異的にペアを作ることができるのかそのメカニズムは謎でした。このため今回、X線共結晶構造解析により相互作用機構の解明に挑みました。
このような直交性RBP−RBPペアは生命科学研究のためのツール開発用の分子パーツとして有用ですが、これまでにCS1−LS4、CS2−LS12を利用した研究はあまり報告されていません。そこで今回、これらRNA−RBPペアを利用して無細胞タンパク質合成系用の遺伝子発現制御ツール(リボスイッチ)の開発を行いました。
研究成果
今回、共同研究グループはX線共結晶構造解析を行い人工RNA−タンパク質複合体(RNP)であるCS1−LS4及びCS2−LS12の構造を決定することに成功しました(図1)。これら二つのRNPは祖先にあたるキンクターンRNA−L7Ae複合体と非常に似通った全体構造をしていることが分かりました。一方で、LS4/LS12タンパク質に変異導入されたアミノ酸残基がπスタックや水素結合など異なる結合モードを介してCS1/CS2 RNAアプタマーを特異的に認識することが明らかとなりました。また、CS1/CS2 RNAはいずれもキンクターン様の構造を取ることが分かりましたが、とりわけCS1 RNAは非バルジ側のループが拡大、またグアニン塩基同士で非ワトソン−クリック塩基対を形成するなど、これまでに構造が解かれている天然のキンクターンRNAには存在しない特徴的な部分構造を持つことを発見しました。さらに、LS12はCS2 RNA非存在下でホモ二量体を形成することを見出しました。二量体化はLS4や祖先タンパク質L7Aeには見られない性質であり、二量体化することで本来のパートナーではない低親和性のキンクターンRNAのアクセスを防ぐという新たなメカニズムが提唱されました。
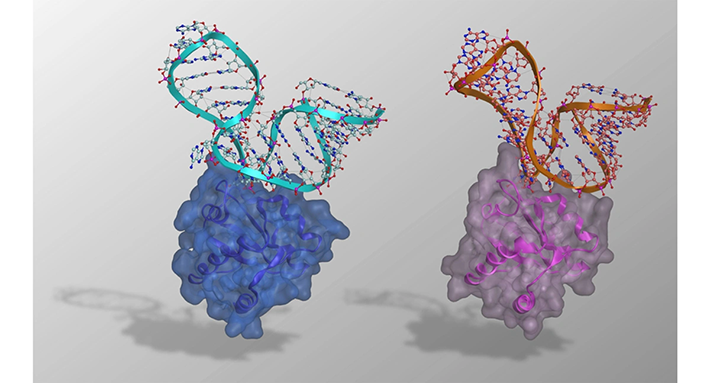
図1. CS1−LS4(左)、CS2−LS12(右)の構造
共同研究グループはさらに、無細胞転写・翻訳システムで機能するタンパク質リガンド応答型のリボスイッチの開発に取り組みました(図2)。T7プロモーター配列の直後にRNAアプタマー配列(CS1、またはCS2)を挿入し、転写時にタンパク質リガンド(LS4、またはLS12)が存在しない条件ではリボソーム認識配列を遮蔽することで、下流の遺伝子の翻訳が抑制されるようスイッチのデザインを行いました。タンパク質リガンドが存在する条件では、転写直後にタンパク質リガンドが結合のパートナーであるRNAアプタマーと複合体を形成することでリボソーム認識配列がオープンになり、リボソームによる翻訳(タンパク質合成)が行われます。つまり、タンパク質リガンドが存在する条件でのみタンパク質合成が行われるONスイッチを作製しました。蛍光タンパク質(EGFP)レポーターの発現レベルを指標にしたスクリーニングにより、ON/OFF比500に匹敵する世界最高性能のセルフリーリボスイッチを同定することに成功しました。これまでに、低分子やタンパク質などのリガンドに応答するセルフリーリボスイッチが報告されてきましたが、ON/OFF比が3桁を超えるものは知られていません。本研究では、3桁のON/OFF比を示す直交性リボスイッチ二組を開発することに成功しています。
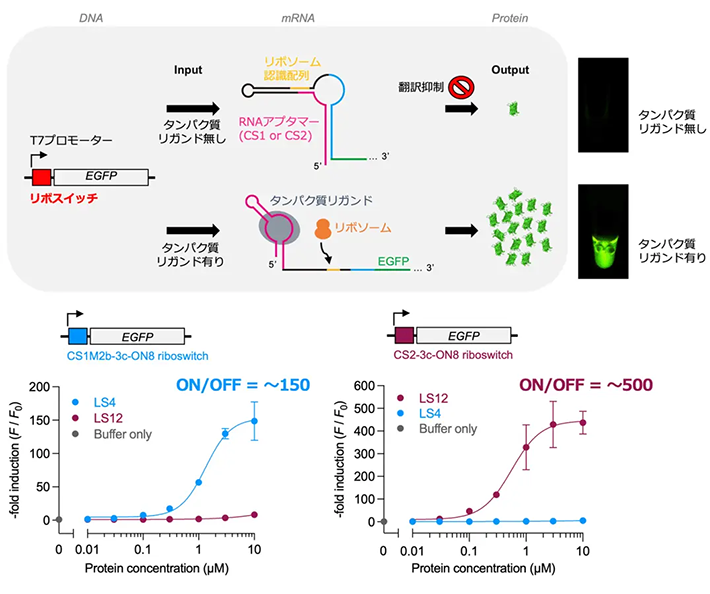
図2. セルフリーシステムを用いたタンパク質応答リボスイッチの評価(論文中の図を改変)
γヘモリシンはHlg2およびLukFから構成される二成分系の膜タンパク質であり、赤血球にナノポアを形成することで溶血を引き起こします。一成分系のαヘモリシンは無細胞(セルフリー)合成生物学、とりわけ人工細胞を用いた研究において利用されることが多い膜タンパク質ですが、γヘモリシンを使った研究例はありませんでした。今回開発した二つのリボスイッチを用い、セルフリー系においてLS4・LS12二つのタンパク質リガンドが存在する条件でのみγヘモリシンが機能発現する論理回路(AND回路)の構築を行いました。ヘモリシンの活性を高感度に検出することが可能なウサギ赤血球を用いて溶血アッセイを行ったところ、LS4・LS12が存在する条件でのみ溶血が観察され、AND回路が機能していることが示されました(図3)。同様に、人工脂質二重膜(LUVs)を用いた実験系においても、AND回路を使ったγ−ヘモリシンの機能発現制御を行うことに成功しています。γヘモリシンがセルフリー系において機能を持つ形で合成できることを示したのは新たな知見です。
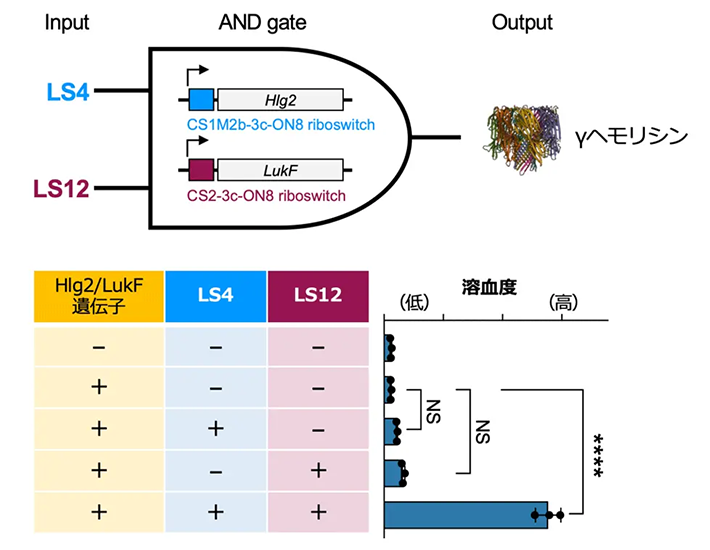
図3. AND回路を用いたγヘモリシンの機能発現制御(論文中の図を改変)
展望
CS1−LS4、CS2−LS12の構造解析によりこれらRNPにおける相互作用の違い、また共通する相互作用が明らかとなりました。今後、RNP間の直交性をさらに向上させるため、分子・ライブラリーのデザインに構造情報が活用されることが期待されます。また今回、CS1−LS4、CS2−LS12を用いることで3桁のON/OFF比を示す高性能セルフリーリボスイッチを開発することに成功しましたが、さらなるON/OFF比の向上を目指し、カスケード回路の構築などを進めています。これらRNA−RBPペアが大腸菌・動物細胞など異なるプラットフォーム(生細胞)で機能するのかも今後の検証課題の一つです。
発表者
- 宮崎大学 テニュアトラック推進室:福永 圭佑(准教授)
- 九州大学 大学院農学研究院:寺本 岳大(助教)、中島 もも香(大学院生)、角田 佳充(教授)
- 東京科学大学 地球生命研究所:福永 圭佑(特任助教, 研究当時)、大谷 俊貴(大学院生)、松浦 友亮(教授)
- 沖縄科学技術大学院大学 核酸化学・工学ユニット:福永 圭佑(ポストドクトラルスカラー, 研究当時)、横林 洋平(教授)
- 電気通信大学 大学院情報理工学研究科:勝木 陸(大学院生)
- 用語説明
[用語1] 無細胞タンパク質合成系(セルフリーシステム):転写に必要なRNAポリメラーゼや翻訳に必要なリボソームなどタンパク質合成に必要な成分を含む反応液であり、DNA/mRNAを追加するだけでタンパク質が合成される。近年、セルフリーシステムを用いた無細胞合成生物学研究が盛んに行われている。
[用語2]
PD-SELEX法:ファージディスプレイライブラリー × RNAライブラリーのセレクションを行う共進化分子工学の手法。タンパク質を提示するファージライブラリーとRNAライブラリーを構築し、試験管内で結合するRNA−RBPペアのみを選択・増幅するという操作を繰り返すことが特徴。下記の論文に詳細が掲載されています。
雑誌名:Nucleic Acids Research(2021年7月5日 オンライン公開)
タイトル:Directed evolution of orthogonal RNA–RBP pairs through library-vs-library in vitro selection
著者:Keisuke Fukunaga, Yohei Yokobayashi*
DOI:10.1093/nar/gkab527![]()
[用語3] 直交性:高い結合選択性、また排他性があること。
- 論文情報
| 掲載誌: | Nucleic Acids Research(2025年3月23日 オンライン公開) |
|---|---|
| タイトル: | Structural insights into lab-coevolved RNA–RBP pairs and applications of synthetic riboswitches in cell-free system |
| 著者: | Keisuke Fukunaga*,#, Takamasa Teramoto#, Momoka Nakashima, Toshitaka Ohtani, Riku Katsuki, Tomoaki Matsuura, Yohei Yokobayashi*, Yoshimitsu Kakuta* #contributed equally, *corresponding authors |
| DOI: | 10.1093/nar/gkaf212 |